在细胞核这个微小的空间中,长达2米的DNA并非杂乱无章地堆砌,而是通过精密的染色质折叠形成复杂的三维结构,这种空间构象直接决定了基因的表达调控。特定蛋白介导的染色质互作捕获技术是研究基因组三维结构和基因调控网络的重要工具,这些技术通过结合蛋白质与DNA的相互作用,解析基因组的远程互作关系,揭示基因表达调控的分子机制。ChIA-PET、HiChIP和PLAC-seq都是用于研究特定蛋白介导的染色质互作的技术,它们在实验流程、灵敏度、成本和应用场景上有一定的相似性和差异,伯小医今天带大家了解下这三种前沿技术,为您探索基因调控的复杂机制提供新的思路和方向。
一、ChIA-PET
背景:
ChIA-PET(染色质互作分析配对末端标签测序,Chromatin Interaction Analysis by Paired-End Tag Sequencing)是一种最早出现的用于研究染色质三维结构和蛋白质介导的DNA互作的技术之一。它结合了染色质免疫沉淀(ChIP)和配对末端标签测序(PET sequencing)的优势,能够在全基因组范围内检测蛋白质介导的染色质相互作用。ChIA-PET技术于2009年首次被开发(Fullwood et al., 2009),旨在解决传统3C(染色体构象捕获)技术及其衍生方法(如4C、5C和Hi-C)在分辨率和功能特异性上的不足。与Hi-C相比,ChIA-PET通过引入ChIP步骤,能够特异性富集目标蛋白质结合的染色质片段,从而提供更高的分辨率和功能特异性。
为了进一步提高分辨率和单倍型特异性,2017年阮一骏和李国亮老师课题组研发了Long-read ChIA-PET(长读长染色质互作分析配对末端标签测序),通过增加测序读长来提高染色质互作分析的分辨率和准确性(Li et al., 2017)。此外还有一些在传统ChIA-PET技术基础上发展而来的改进版本技术也在陆续出现,如受到in situ Hi-C方法的启发,通过在细胞核内进行染色质的消化和连接反应,保留染色质的三维结构发展的in situ ChIA-PET(原位ChIA-PET)技术,该方法减少了操作过程中的结构破坏,降低随机连接噪音,能够更高效地捕获染色质互作(Wang et al., 2021)。
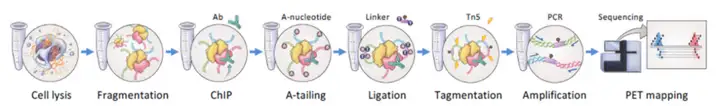
技术原理(Long-read ChIA-PET):
(1)染色质交联与固定。使用甲醛(Formaldehyde)和乙二醇双琥珀酰亚胺(Ethylene glycol bis succinimide,EGS)对细胞进行双重交联,固定染色质中蛋白质与DNA的相互作用。
(2)染色质免疫沉淀(ChIP)。细胞和核膜裂解后,通过超声波将染色质片段化,随后使用特异性抗体富集目标蛋白质结合的染色质复合物。
(3)邻近连接反应。进行DNA末端修复和A-tailing。在DNA片段末端添加“桥接连接子”(bridge linker),通过邻近连接反应将相互靠近的DNA片段连接在一起,随后DNA通过解交联释放出来。
(4)Tn5转座酶处理。使用Tn5转座酶随机切割邻近连接后的染色质DNA片段,同时在DNA末端加入测序接头。
(5)片段捕获及扩增。使用链霉亲和素磁珠捕获含生物素的片段,并进行PCR及测序分析。
技术优势:
(1)全基因组范围:能够在全基因组水平上检测蛋白质介导的染色质相互作用。
(2)高分辨率:通过连接和酶切步骤,能够精确识别相互作用的DNA区域。
(3)特异性高:结合ChIP步骤,能够特异性富集目标蛋白质结合的DNA片段。
(4)适用于多种蛋白质:可以用于研究转录因子、组蛋白修饰、染色质重塑因子等多种蛋白质介导的染色质互作。
技术应用:
(1)基因组三维结构解析:在全基因组范围内检测染色质的远程相互作用,揭示基因组的三维折叠方式,解析肿瘤细胞中的染色质相互作用,在疾病相关的基因组结构研究中发挥重要作用。
(2)转录调控网络研究:广泛应用于研究转录因子、辅因子和组蛋白修饰介导的染色质相互作用。
(3)增强子-启动子互作分析:能够将基因与它们的顺式调控元件(如增强子)连接起来,揭示基因表达调控的分子机制。
文章案例:
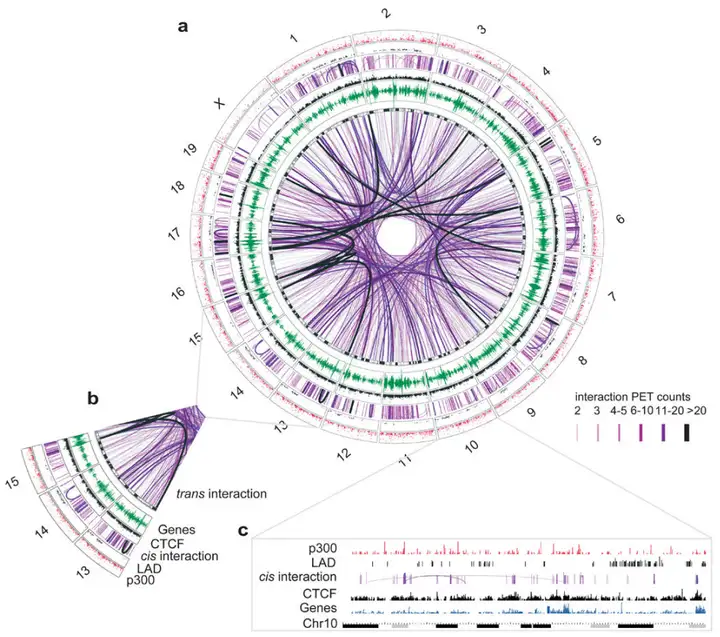
Handoko等人利用ChIA-PET技术研究了小鼠胚胎干细胞中CTCF介导的染色质互作网络,揭示了CTCF在基因组三维结构和转录调控中的作用。
(1)鉴定CTCF结合位点和染色质互作:通过ChIA-PET技术,研究者鉴定了39,371个CTCF结合位点和1,816个高置信度的染色质互作位点(包括1,480个顺式互作和336个反式互作),这些互作位点在基因组中分布广泛,涉及多个基因。并通过4C技术和荧光原位杂交(FISH)验证了ChIA-PET鉴定的染色质互作,证实了CTCF在染色质互作中的关键作用。
(2)CTCF在染色质结构中的作用:CTCF通过形成染色质环,将基因组划分为不同的功能区域。这些环可能作为“域屏障”,限制异染色质的扩散。CTCF结合位点倾向于位于核膜附着区域的边界,可能通过限制异染色质的扩散来维持基因表达的调控。
(3)染色质互作与基因调控:CTCF介导的染色质互作能够将增强子与启动子连接起来,促进基因表达。通过分析CTCF敲低对基因表达的影响,研究者发现CTCF介导的染色质互作对基因表达具有重要的调控作用。
二、HiChIP
背景:
HiChIP(in situ Hi-C followed by chromatin immunoprecipitation)技术由斯坦福大学Howard Chang实验室于2016年开发,并在《Nature Methods》上发表(Mumbach et al., 2016)。HiChIP融合了Hi-C和ChIA-PET技术的优势,并借助转座酶介导的文库构建方法,在较低的数据量下实现更高分辨率的染色质三维结构解析。HiChIP技术凭借其高效、灵敏和低样本需求的特点,已成为研究染色质三维结构和基因调控机制的重要工具。
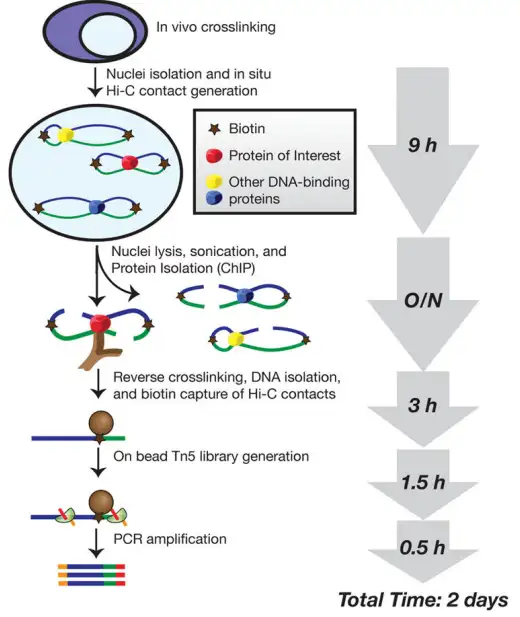
技术原理:
HiChIP的核心在于将Hi-C的原位交联与ChIP-seq的特异性富集相结合。首先对细胞进行交联,然后收集细胞核并产生原位Hi-C交联。将细胞核裂解,对Hi-C连接产物进行片段化打断,利用特异性抗体进行免疫共沉淀ChIP富集,解交联后分离DNA,利用生物素捕获互作序列进行建库测序。
技术优势:
HiChIP技术相比传统Hi-C和ChIA-PET技术具有显著优势:
(1)更低的样本需求:相比传统的ChIA-PET技术,HiChIP的样本需求降低了100倍以上,细胞数量要求也低于Hi-C。
(2)更高的灵敏度:HiChIP能够在更小的数据量下获取更高分辨率的染色质构象信息,显著提高了检测效率。
(3)更高的信噪比:通过原位交联和特异性富集,HiChIP显著降低了假阳性,提高了数据的可靠性。
数据库与资源:
2022年,斯坦福大学Wing Hung Wong教授课题组与清华大学江瑞课题组联合发布了首个附带全基因组功能注释的HiChIP数据库(HiChIPdb),收录了超过200个人类HiChIP样本的数据(Zeng et al., 2023)。该数据库为研究人员提供了丰富的资源,用于探索基因调控网络和染色质构象的变化。
三、PLAC-seq
背景:
PLAC-seq(Proximity Ligation-Assisted ChIP-Seq)技术是由加州大学圣地亚哥分校的于淼博士在博士后期间开发的一种用于分析染色质三维结构和长程相互作用的高通量测序技术。该技术的开发背景是基于对现有染色质构象捕获技术(如Hi-C和ChIA-PET)的改进需求,这些技术虽然能够提供全基因组范围内的染色质相互作用信息,但存在样本需求量大、测序成本高以及操作复杂等局限。PLAC-seq通过调整实验步骤的顺序,将邻近连接反应提前至染色质片段化之前,显著提高了检测效率和灵敏度(Fang et al., 2016)。
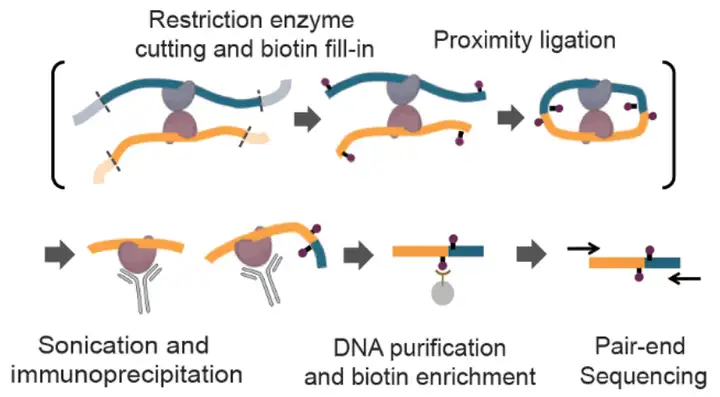
技术优势:
(1)低样本需求:PLAC-seq显著降低了样本需求量,相比ChIA-PET,其细胞输入量减少。
(2)高分辨率:能够在千碱基(kilobase)级别分辨率下检测长程染色质相互作用。
(3)高灵敏度:通过优化实验步骤,PLAC-seq在检测染色质相互作用方面表现出更高的灵敏度和准确性。
(4)成本效益:相比Hi-C和ChIA-PET,PLAC-seq显著降低了测序成本。
技术应用:
2024年,任兵、胡明和于淼团队利用PLAC-seq技术靶向H3K4me3组蛋白修饰,揭示了小鼠胎儿发育过程中转录调控相关染色质三维结构的动态变化。该研究在多种小鼠胎儿组织中检测到大量以启动子为中心的染色质远程互作对,这些互作显著富集在启动子与增强子元件之间(Yu et al., 2024)。
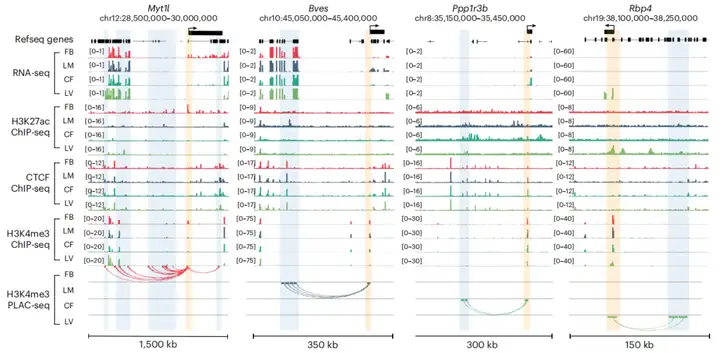
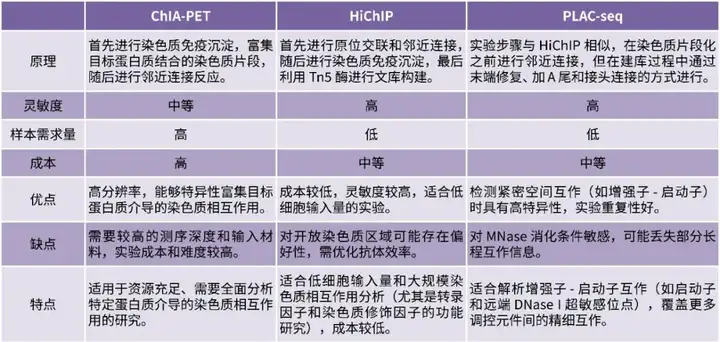
四、技术比较
参考文献
[1] Fullwood M J, Liu M H, Pan Y F, et al. An oestrogen-receptor-α-bound human chromatin interactome[J]. Nature, 2009, 462(7269): 58-64.
[2] Li X, Luo O J, Wang P, et al. Long-read ChIA-PET for base-pair-resolution mapping of haplotype-specific chromatin interactions[J]. Nature protocols, 2017, 12(5): 899-915.
[3] Wang P, Feng Y, Zhu K, et al. In Situ Chromatin Interaction Analysis Using Paired‐End Tag Sequencing[J]. Current protocols, 2021, 1(8): e174.
[4] Mumbach M R, Rubin A J, Flynn R A, et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture[J]. Nature methods, 2016, 13(11): 919-922.
[5] Zeng W, Liu Q, Yin Q, et al. HiChIPdb: a comprehensive database of HiChIP regulatory interactions[J]. Nucleic acids research, 2023, 51(D1): D159-D166.
[6] Fang R, Yu M, Li G, et al. Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq[J]. Cell research, 2016, 26(12): 1345-1348.
[7] Yu M, Zemke N R, Chen Z, et al. Integrative analysis of the 3D genome and epigenome in mouse embryonic tissues[J]. Nature Structural & Molecular Biology, 2024: 1-12.
[8] Handoko L, Xu H, Li G, et al. CTCF-mediated functional chromatin interactome in pluripotent cells[J]. Nature genetics, 2011, 43(7): 630-638.

