今日,《自然》杂志及旗下《自然·医学》杂志刊发了一系列TRACERx计划新结果,这是一项持续收集非小细胞肺癌(NSCLC)患者综合基因组数据和临床数据的大型研究项目,从诊断到治疗全面揭秘NSCLC,对NSCLC的自然史提供了最全面的描述。共计7篇论文包括肺癌如何进化、扩散、对治疗耐药、预测预后等等主题,实在是非常精彩。
先来说说什么是TRACERx。
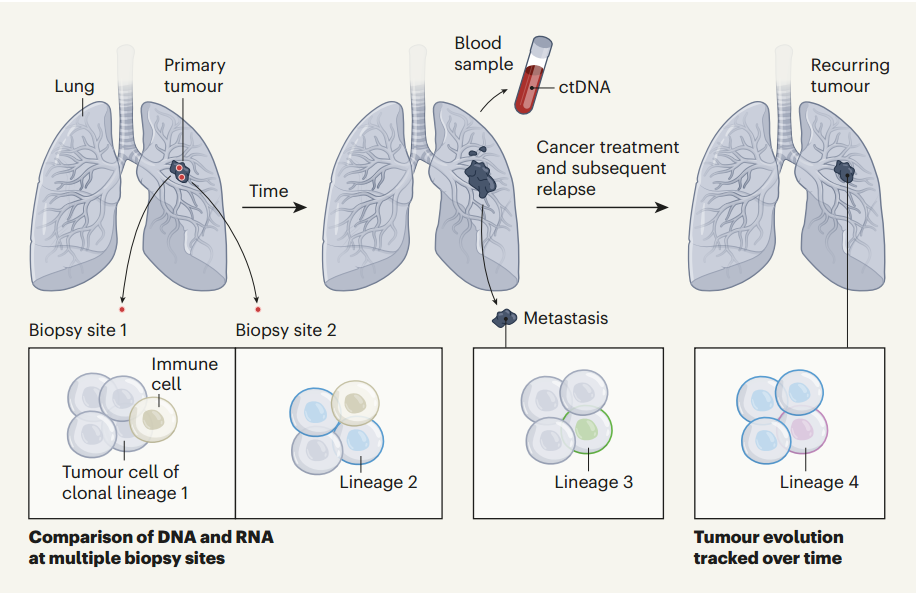
9年、14万英镑、19个位于英国的中心、250名研究人员、800余名NSCLC患者——这是英国癌症研究中心(CANCER RESEARCH UK)给出的一组与TRACERx有关的数字。TRACERx成立于2014年,整合多种技术,分析肿瘤DNA的纵向演变,这包括从血液中检测这些变化、表征肿瘤免疫和组织微环境、破译癌症进化模式和机制等等,并尝试从中找到治疗癌症的新方法。
研究者发现在肺腺癌中,40个常见癌基因中的22个有显著的选择优势,包括经典癌相关基因TP53和KRAS。有趣的是,在8%有吸烟史的患者中,却不存在烟草相关突变,而且与不吸烟的患者样本相比,它们EGFR、ROS1、ALK、MET等突变检出率也很相似,这说明它们具有相似的病因和发病机制。
亚克隆扩增与选择优势有关,在肿瘤发育较晚时间点出现亚克隆扩增的患者无病生存期明显缩短。
19%的肿瘤中存在全基因组倍增(WGD)事件,10%肿瘤同时存在多个WGD,WGD与较短的无病生存期有关。
分析结果显示,25%的病例中转移灶分化处于较早阶段,而且诊断时正在吸烟的患者早期分化更为丰富。值得关注的是,对原发瘤的单一区域采样导致有多达83%的晚期分化病例被错误分类为早期,这突出了多点取样的重要性。在32%的病例中发现了与胸外复发相关的多克隆传播。原发淋巴结疾病导致的转移占比不到20%。导致转移的亚克隆在原发瘤内出现扩增,可能是正向选择的结果。
这项研究强调了在原发瘤中亚克隆对转移的作用、对转移区域的影响、当前筛查方法的局限性。
#以上两篇文章的通讯作者都是PM2.5致癌这篇论文的通讯,想问问大佬两周发3篇nature是啥感觉……
#其实他们还发了一篇nature medicine下面讲
对代表原发瘤和转移瘤的947个区域和96个邻近正常组织样本的分析结果显示,转录组是肿瘤表型变异的主要原因。分析中频繁观察到拷贝数依赖性等位基因特异表达,这与表观遗传变异有关。
研究者还结合多种机器学习方法,将转移潜力与原发瘤内突变的进化背景进行练习。这些分析强调了肿瘤异质性、进化和转移背后的基因机制。
研究发现,任何小鼠肺腺癌都会引起局部生发中心反应和抗肿瘤抗体,并鉴定到内源性逆转录病毒(ERV)包膜糖蛋白是抗体的主要靶标,这依赖于CXCL13依赖性TLS。这种反应能被免疫治疗放大,并与KRAS抑制形成协同效应。
ERV反应性抗体能够发挥抗肿瘤活性,延长荷瘤小鼠生存期,而且人类患者中ERV的表达也能预测免疫治疗预后。
研究者们通过一种名为a tumor-informed personalized cell-free DNA enrichment approach的方法,富集循环肿瘤DNA(ctDNA)并进行微小残留病(MRD)检测,灵敏性和特异性令人惊叹,可检测低至总DNA 0.003%的变异。
在复发的45名患者中,有37名检测到ctDNA,而随访期间发生第二原发癌的10名患者均未检测到ctDNA,说明特异性还是不错的。
至于新论文里有什么新内容,《自然》你倒是赶紧把论文上线啊!!
对651名患者使用计算机断层扫描进行的身体成分分析显示,在诊断时骨骼肌或脂肪分布最少的20%患者,生存期明显缩短。这个发现在另外的队列中也得到了验证。
研究者发现,出现CAC的患者具有特定的肿瘤基因组和转录组,包括炎症信号和上皮-间充质转换途径的富集,以及MAGEA6、ADAMTS3等基因的上调。
进一步分析发现,生长分化因子(GDF15)与体重、骨骼肌和脂肪组织减轻之间存在显著关联,或许能够成为CAC管理的靶点。
肺腺癌有多种组织学形态,但对形态如何反映肿瘤的进化和疾病进展我们还知之甚少。
这项研究[7]匹配了原发瘤、配对转移样本的外显子组和DNA测序及组织病理学分析,发现高级别肿瘤表现出较高的增殖和较低的克隆多样性。不同组织学特征的肿瘤可见不同的基因频率,表明存在不同的进化轨迹。研究还发现不同形态与复发位置存在关联。
随着这些发现的公布,TRACERx的第一阶段目标基本完成,接下来,研究者们即将开展名为TRACERx EVO的下一阶段计划,将在TRACERx发现的基础上继续深入挖掘肺癌背后的故事,为临床诊疗带来新变革。
参考资料:
[1]Frankell, A.M., Dietzen, M., Al Bakir, M. et al. The evolution of lung cancer and impact of subclonal selection in TRACERx. Nature (2023). https://doi.org/10.1038/s41586-023-05783-5
[2]Al Bakir, M., Huebner, A., Martínez-Ruiz, C. et al. The evolution of non-small cell lung cancer metastases in TRACERx. Nature (2023). https://doi.org/10.1038/s41586-023-05729-x
[3]Martínez-Ruiz, C., Black, J.R.M., Puttick, C. et al. Genomic–transcriptomic evolution in lung cancer and metastasis. Nature (2023). https://doi.org/10.1038/s41586-023-05706-4
[4]Ng, K.W., Boumelha, J., Enfield, K.S.S. et al. Antibodies against endogenous retroviruses promote lung cancer immunotherapy. Nature (2023). https://doi.org/10.1038/s41586-023-05771-9
[5]Abbosh C, Frankell A, Garnett A, et al. Phylogenetic tracking and minimal residual disease detection using ctDNA in early-stage NSCLC: A lung TRACERx study. AACR Virtual Meeting; April 27-28, 2020. Abstract CT023.
[6]Al-Sawaf, O., Weiss, J., Skrzypski, M. et al. Body composition and lung cancer-associated cachexia in TRACERx. Nat Med (2023). https://doi.org/10.1038/s41591-023-02232-8
[7]Karasaki, T., Moore, D.A., Veeriah, S. et al. Evolutionary characterization of lung adenocarcinoma morphology in TRACERx. Nat Med (2023). https://doi.org/10.1038/s41591-023-02230-w
文章来源:奇点网