在基于质谱的蛋白质组学技术中,其核心技术主要包括蛋白质组学定性和定量两大类,其中定量蛋白质组学是蛋白质组学核心技术中的核心。现阶段最主流、商业化应用最广的蛋白质组学方法包括基于质谱数据依赖型采集模式(Data-dependent acquisition,DDA)也称为“鸟枪法”(Shotgun)的同位素标记相对和绝对定量技术(Isobaric tags for relative and absolute quantification,iTRAQ)、串联质谱标记技术(Tandem mass tag,TMT)、细胞培养条件下稳定同位素标记技术(Stable-isotope labelling by amino acids in cell culture,SILAC)、基于非标记定量技术(Label-free)以及基于质谱数据非依赖型采集模式(Data-independent acquisition)的DIA/SWATH技术。
原理简介
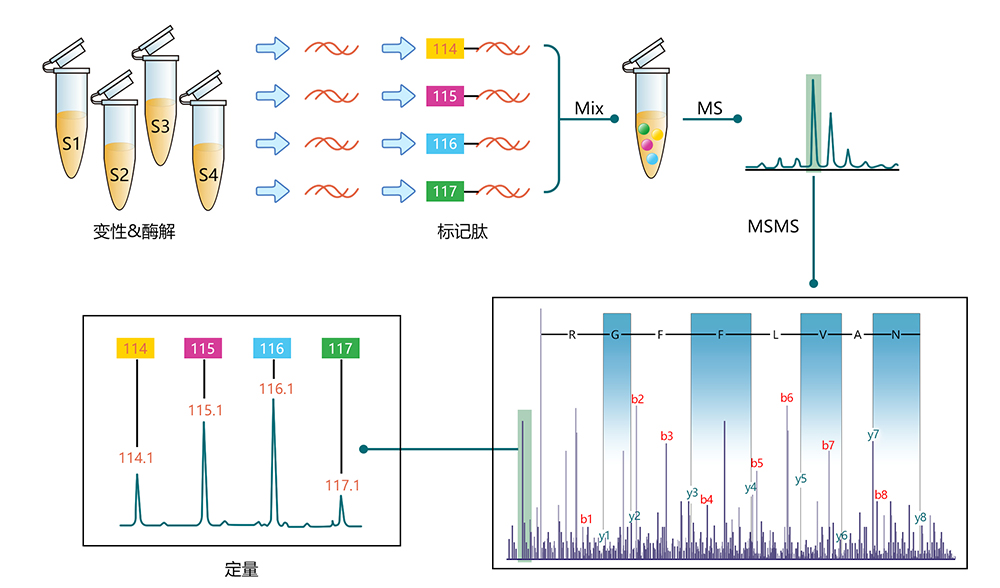
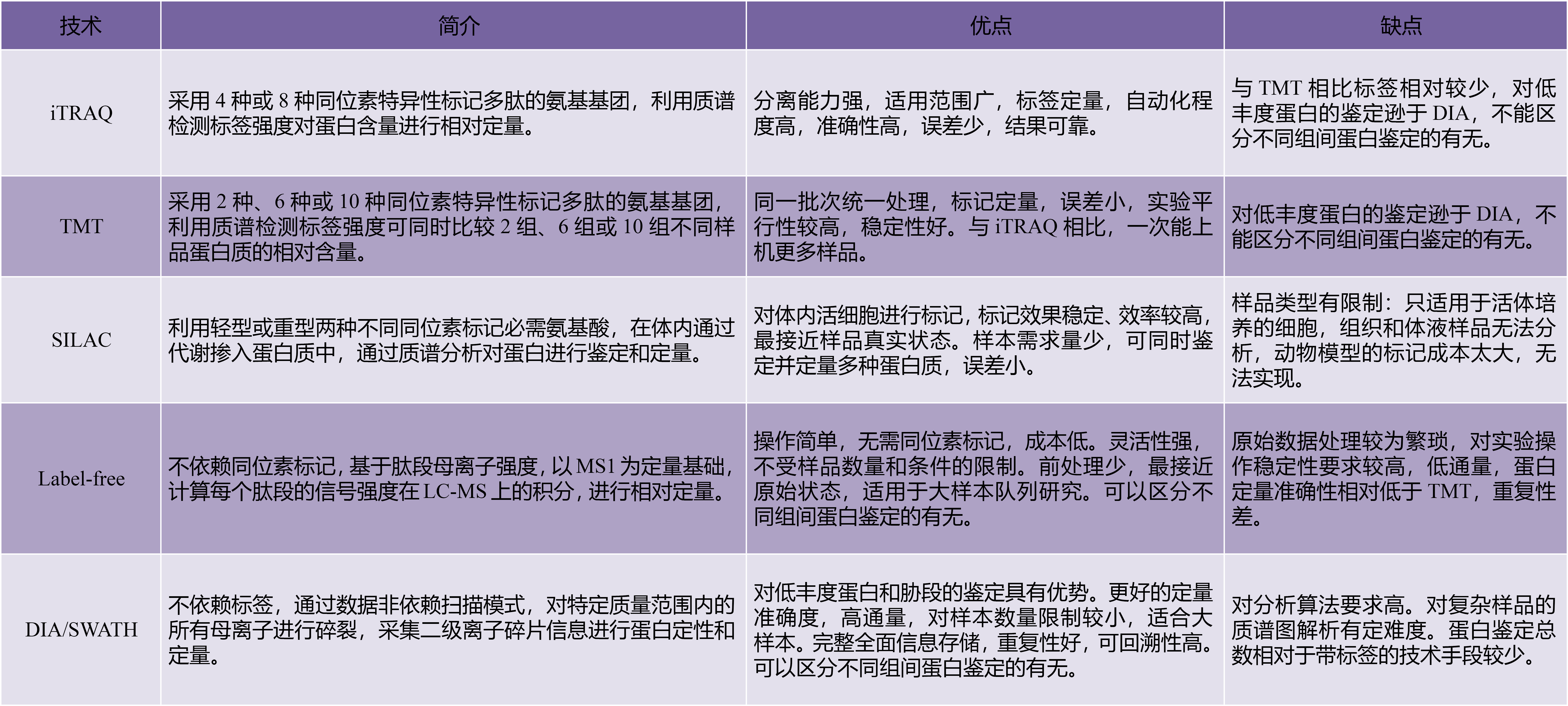
iTRAQ技术采用4种或8种同位素编码的标签,通过对氨基酸N端和赖氨酸残基进行酶解后用iTRAQ试剂进行特异性标记,而后进行串联质谱分析,可同时比较4种或8种不同样品中蛋白质的相对含量。
iTRAQ试剂包括3个部分:报告基团(Report group)、平衡基团(Balance group)和肽反应基团(Amine-specific reactive group)。报告基团相对分子质量为113-121Da(无120),因此iTRAQ试剂可同时标记8组样品。平衡基团相对分子质量为192-184Da(无185),使得八种iTRAQ试剂分子量均为305Da,保证标记的同一肽段质荷比(m/z)相同。肽反应基团将iTRAQ试剂与肽段的N端及赖氨酸的氨基特异结合,在其上报告基团通过平衡基团与反应基团相连,形成等量异位标签。
在串联质谱中,来自不同样品的同一肽段由于是等质量的,在一级质谱检测(MS1)中表现为一个母离子峰(即同一个质谱峰),在二级质谱检测(MS2)中进一步碎裂时,报告基团、质量平衡基团和多肽反应基团之间的键断裂,质量平衡基团丢失,报告基团则会产生相应的不同质量数的报告离子,它们各自质谱峰的信号强弱,代表着来源于不同样品的该肽段及其所对应的蛋白的表达量的高低。因此,根据报告离子的信号强度值即可获得样品间相同肽段的定量信息,再经过软件处理得到蛋白质的定量信息。同时,多肽内的酰胺键断裂,形成一系列b离子和y离子,得到离子片段的质量数,通过数据库查询和比较,可以鉴定出相应的蛋白质前体。
实验流程
蛋白提取和质控——iTRAQ标记肽段——HPLC分级——质谱分析——数据库处理——生物信息学分析
原理简介
TMT技术采用2种、6种、10种或16种同位素标签,特异性标记多肽的氨基基团,然后进行串联质谱分析,监测碎裂下来的标签实现肽段定量。一次实验可同时比较最多16组不同样品中蛋白质的相对含量。
TMT标签由三部分组成:分子量报告子(报告基团)、分子量标准化部分(平衡基团)和(肽)反应基团,形成2种、6种、10种或16种相对分子质量均为等量的异位标签。TMT试剂通过反应基团与肽段上赖氨酸及N端氨基酸残基相连,能够高效地标记酶解后的肽段。在一级质谱中,分子量标准化部分使任何一种TMT试剂标记的不同样本中的同一肽段表现为相同的质荷比。在二级质谱中,释放出分子量报告子。每个报告子都有各自独特的分子量,产生不同的报告离子峰,其强度反应了该肽段在不同样品中的相对表达量信息,另外二级质谱中的肽段碎片离子峰质荷比反应了该肽段的序列信息,根据这些质谱原始数据可得到蛋白质的定性和相对定量信息。
实验流程
蛋白提取和质控——TMT标记肽段——HPLC分级——质谱分析——数据库处理——生物信息学分析
最初SILAC使用的标记氨基酸是氚代甲硫氨酸和氘代甘氨酸,目前常用的标记氨基酸有亮氨酸、精氨酸、赖氨酸、甲硫氨酸和酪氨酸等。
原理简介
在细胞培养基中加入天然同位素(轻型)或稳定同位素(重型)标记的必需氨基酸,替代细胞培养基中相应的氨基酸,细胞经若干传代后,稳定同位素标记的氨基酸完全掺入到细胞新合成的蛋白质中,取代了原有的氨基酸。这样,两个蛋白之间就存在分子量的改变,而其它化学性质无异。然后将两组细胞混合,提取蛋白质,经分离纯化后进行质谱测定。
每个肽段作为质谱中的一对:低分子量的肽段含有轻型氨基酸,来源于A组,高分子量的肽段则含有重型氨基酸,来源于B组。如果SILAC肽段对呈现1:1的比例,则蛋白质组中此蛋白的丰度无差异。如果含有重型氨基酸的肽段峰强度偏高,则说明B组中蛋白丰度更高。因为稳定同位素标记的氨基酸与天然氨基酸的化学性质基本相同,除了分子量有差异。因此,质谱上峰强度的比值直接对应了A组与B组的比例,即可对蛋白质进行相对定量。
实验流程
细胞培养标记——蛋白提纯——1:1混合蛋白样品——蛋白还原、酶切——LC-MS液质串联检测——生物信息学分析
原理简介
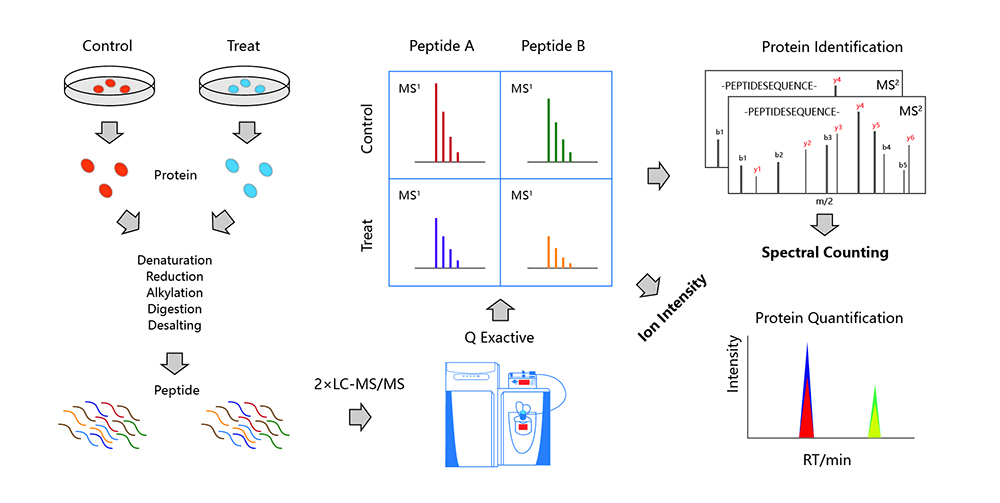
Label-free通过液质联用技术对蛋白质酶解肽段进行质谱分析,按照其原理主要分为两种,第一种Spectrum counts类的非标记方法,发展比较早,已经形成多种定量算法。其主要的原理是以MS2的鉴定结果为定量基础,各种方法的差别在于后期算法在大规模数据上的修正。第二种非标记定量的原理是以MS1为基础,计算每个肽段的信号强度在LC-MS上的积分(如Maxquant),以积分面积对肽段对应的蛋白质进行相对定量。
实验流程
蛋白提取——蛋白酶解——高效液相HPLC预分离——MS质谱分离——原始数据搜库分析——生物信息学分析
DIA采集模式将质谱整个全扫描范围分为若干个小窗口,然后依次对每个窗口中的所有离子进行检测、碎裂,使其能够对扫描区间内的所有肽段离子进行高速一级MS扫描,再进行二级MS/MS分析,从而无遗漏、无差异地获得样本中所有离子的信息,在碎片离子层面完整重现nanoLC的色谱峰图,因此DIA定量可以利用碎片离子随时间的积分来表征肽段含量,而利用二级碎片离子定量不仅可以降低样本检测的缺失值,同时提高定量准确性和重复性,实现大样本队列中高覆盖率,高稳定,高精准的蛋白质组定量分析。但本质上,DIA也是一种Label-free定量方法。
原理简介
DIA/SWATH技术将扫描范围划分为以25 Dalton为间隔的一系列区间,通过超高速扫描来获得扫描范围内全部离子的所有碎片信息。以蛋白质组学样品分析中常见的扫描范围400~1200为例,每25 Dalton作为一个扫描间隔(SWATH),每个SWATH扫描时间设定为100 ms,那么该扫描范围累计需要32个SWATH(1200-400/25=32),完成一次扫描仅需要3.2秒。
与传统的shotgun技术相比,SWATH技术能够将扫描区间内所有的肽段母离子经过超高速扫描并进行二级碎裂,使用二级碎片离子进行蛋白相对/绝对定量,从而获得完整的肽段信息。
目前SWATH可以根据样品TIC分布进行窗口优化,从而获取更多的谱图信息,提高了精准度和分辨率。通过高分辨高灵敏质谱TripleTOF 5600/+以及最新的TripleTOF 6600进行分析扫描。因此,SWATH技术是一种真正全景式的、高通量的质谱技术,同时也解决了shotgun鉴定重复度较低的缺点。
实验流程
蛋白提纯——蛋白还原、酶切——HPLC分离肽段——LC-SWATH模式质谱检测——OpenMS分析质谱数据——生物信息学分析
Collins B C, Hunter C L, Liu Y, et al. Multi-laboratory assessment of reproducibility, qualitative and quantitative performance of SWATH-mass spectrometry[J]. Cold Spring Harbor Labs Journals, 2016(1).
Gillet L C, Navarro P, Tate S, et al. Targeted data extraction of the MS/MS spectra generated by data-independent acquisition: a new concept for consistent and accurate proteome analysis.[J]. Molecular & cellular proteomics : MCP, 2012,11(6).
Meyer J. Fast Proteome Identification and Quantification from Data-Dependent Acquisition–Tandem Mass Spectrometry (DDA MS/MS) Using Free Software Tools[J]. Methods & Protocols, 2019, 2(1).
Neilson K A, Ali N A, Muralidharan S, et al. Less label, more free: approaches in label-free quantitative mass spectrometry.[J]. Proteomics, 2011,11(4).
Ross P L, Huang Y N, Marchese J N, et al. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents.[J]. Molecular & cellular proteomics: MCP, 2004,3(12).
Sandra, Isabel, Anjo, et al. SWATH-MS as a tool for biomarker discovery: From basic research to clinical applications[J]. Proteomics, 2017.
Sidoli S, Fujiwara R, Garcia B A. Multiplexed data independent acquisition (MSX-DIA) applied by high resolution mass spectrometry improves quantification quality for the analysis of histone peptides.[J]. Proteomics, 2016,16(15-16).
Thompson A, Schaefer J, Kuhn K, et al. Tandem Mass Tags: A Novel Quantification Strategy for Comparative Analysis of Complex Protein Mixtures by MS/MS[J]. Analytical Chemistry, 2006,78(12).