在基因组DNA没有发生改变的情况下,研究基因功能发生可遗传的变化并最终导致表型变化的原因是表观遗传学的主要内容,其中许多与复杂性状相关的遗传变异优先位于易接近的染色质区域(染色质可及性或开放性)。ATAC-seq是2013年由斯坦福大学William J. Greenleaf和Howard Y. Chang实验室开发的用来研究染色质可及性的方法。与具有同种功能的DNase-Seq、MNase-seq和FAIRE-Seq相比起来,ATAC-seq操作简单,重复性好,实验只需要很少的起始细胞/组织量(<50000个细胞),信号噪比高。与ChIP-seq、CUT&tag以及DAP-seq相比,并不需要有明确的转录因子或组蛋白修饰位点等。ATAC-seq在研究植物生长发育、组织分化和环境响应方面有着广泛的应用前景。今天的内容主要就是给大家介绍ATAC-seq在植物中应用的一些文献实例。别走开,正文马上就要开始啦!

期刊:Postharvest Biology and Technology
发表时间:2022.5.26
影响因子:6.751
技术手段:ATAC-seq、RNA-seq、亚细胞定位、Y1H、双荧光素酶报告基因实验、ChIP-qPCR、EMSA
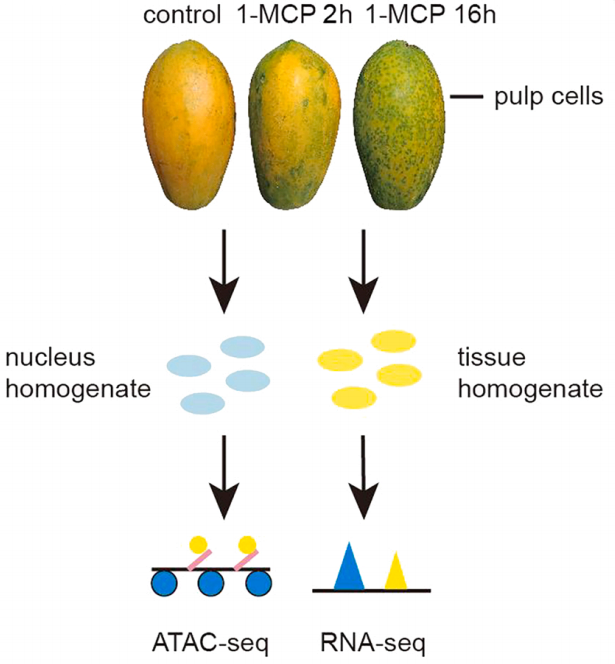
研究人员使用ATAC-seq和RNA-seq联合分析来揭示在木瓜成熟过程中用1-MCP处理不同时间的染色质可及性的差异,旨在确定1-MCP处理下延迟和导致木瓜异常成熟的关键差异可及性区域(Differentially accessible regions,DARs)、上游motif和转录因子(TFs)。这有助于确定在与木瓜成熟相关的乙烯和生长素信号通路的相互作用中起调节作用的基因,这些基因可作为提高水果质量和采后保质期的候选基因(Cai et al., 2022) 。
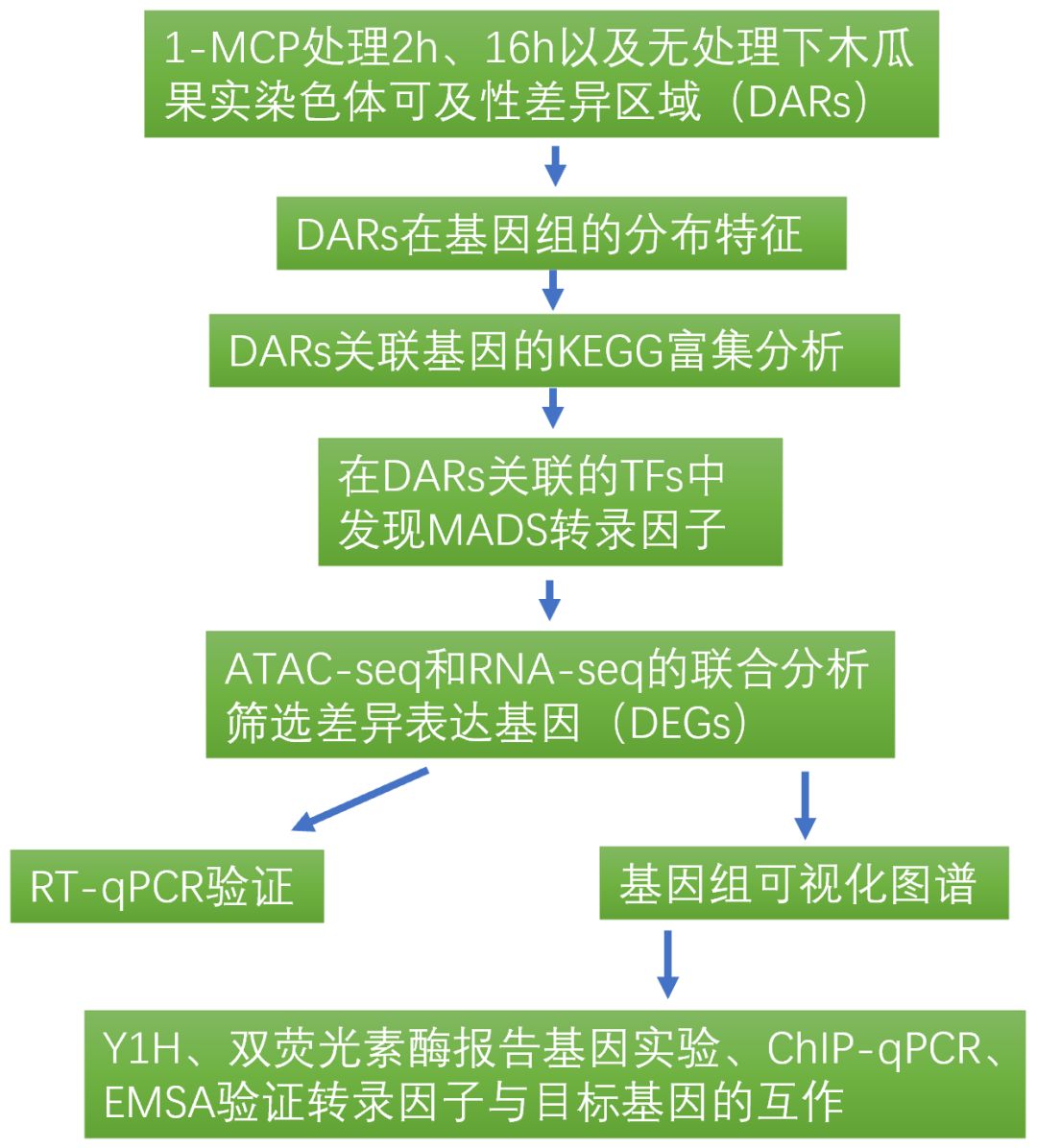
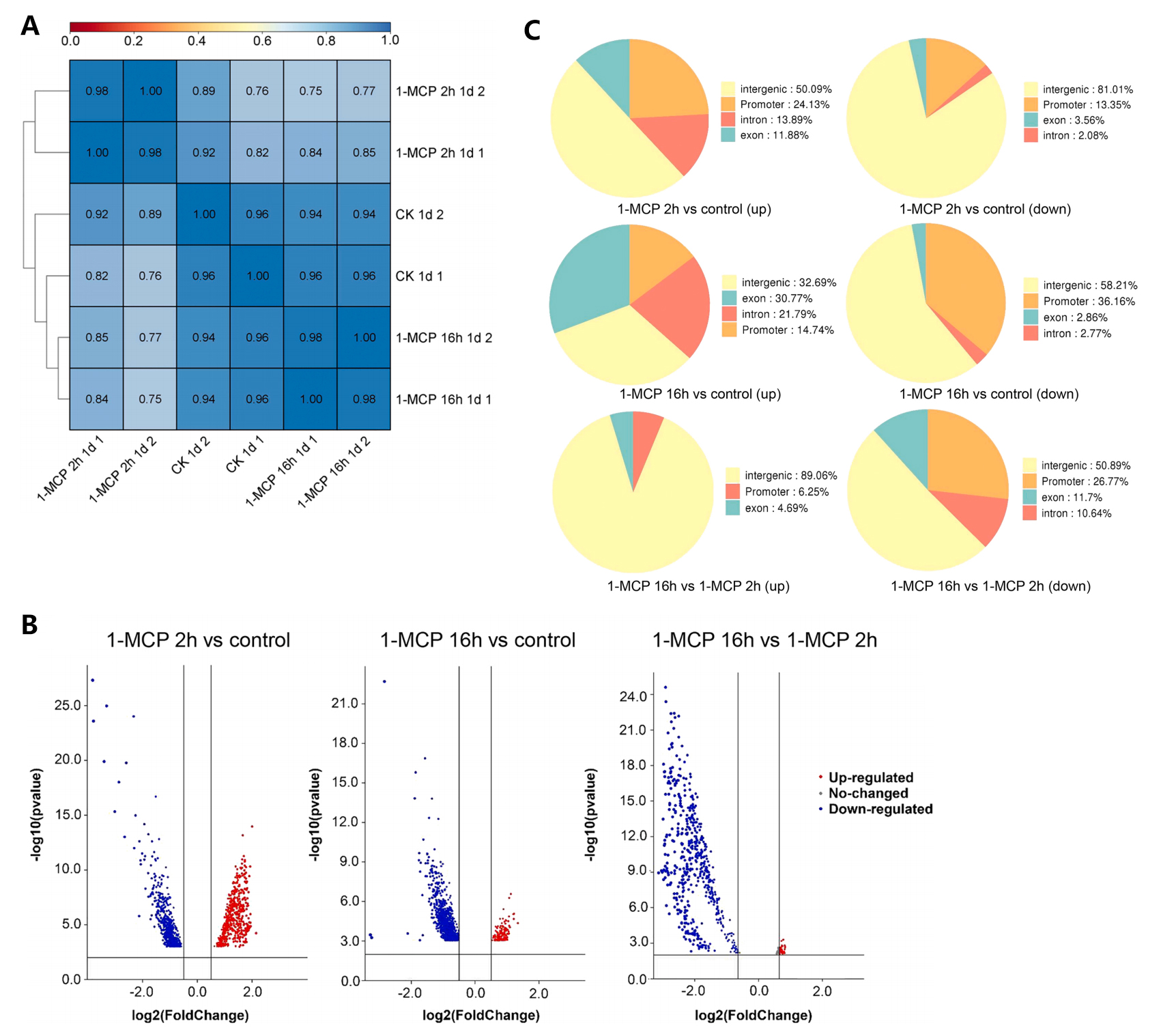
图2 (A)基于ATAC-seq reads数的Pearson相关性聚类热图;(B)1-MCP不同处理下染色质的差异可及性区域(DAR)的火山图。FDR≤0.05且倍数变化≥1.2或≤0.8表示DAR显著。红色圆圈表示上调的DARs,蓝色圆圈表示下调的DARs;(C)不同处理的木瓜果实样品之间DARs在基因组区域(启动子、内含子、编码外显子和远端基因间区域)内的分布情况。
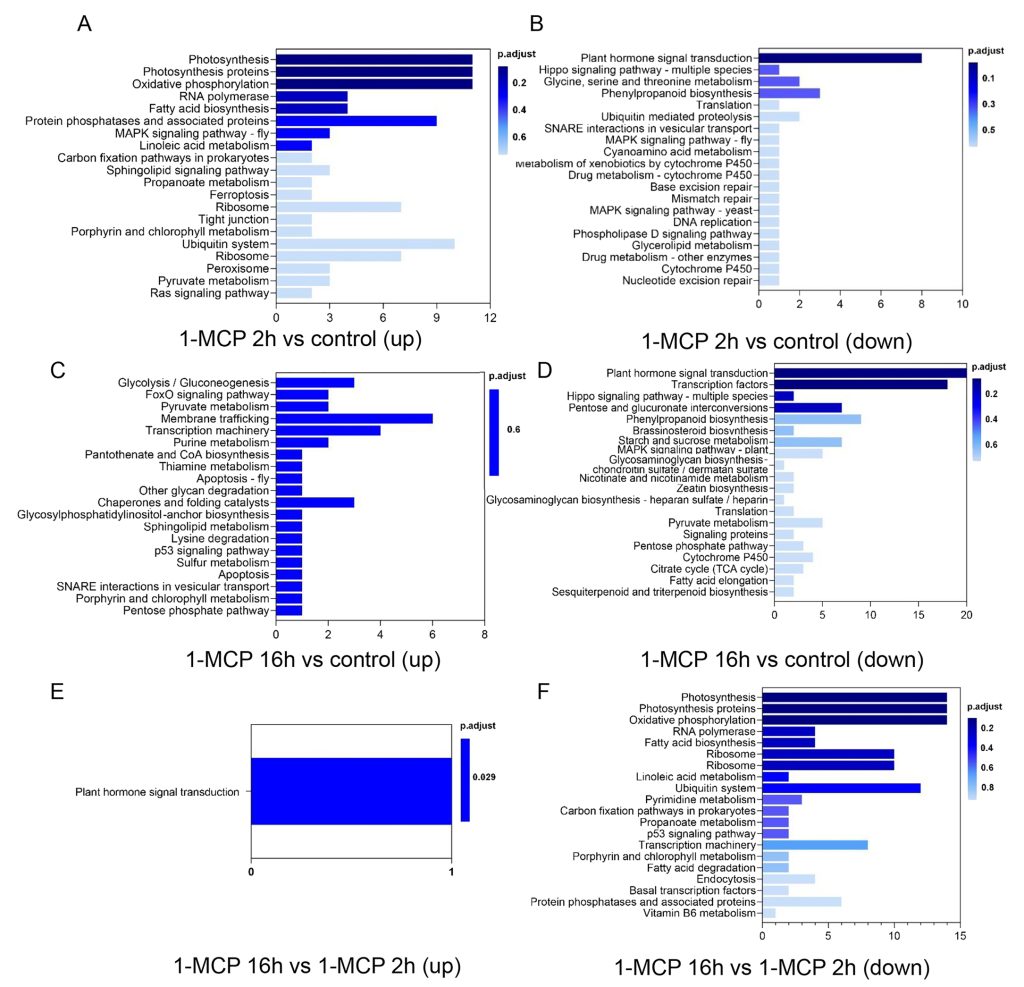
对1-MCP处理上调或下调DARs关联基因的KEGG富集分析表明,植物激素信号转导在1-MCP阻碍果实的成熟和软化中具有重要意义。
图3 1-MCP不同处理下木瓜果实DARs关联基因的KEGG富集bar图。
1-MCP不同处理中DARs关联的转录因子(TF)数据显示,与对照相比,1-MCP处理2h和16h的样品中下调的TFs数量远多于上调的TFs。其中,MADs-M型TFs在1-MCP 2h与对照组相比下调的TFs中占大多数。而在1-MCP处理16h和2h相比上调的TFs中,MADs-M型是唯一差异表达的TFs。在成熟过程中,不同的1-MCP处理显著影响了其他TFs,如MYB、NAC和AP2/ERF-ERF。这些结果表明,MADS-M型转录因子(又称MADS-box转录因子)在1-MCP处理下的木瓜成熟过程中发挥了主要作用(表1)。
已有研究表明,MADS作为一种重要的转录因子,通过多种机制调控植物的成熟。在番茄中,SlMBP9(MADS-box基因之一)调节PIN并减少生长素运输,调节顶端优势和侧根形成,而SlAGL15调节ARF6/8以参与生长素相关信号(Li et al., 2019) 。在木瓜中,CpMADS1/3调节CpEIL1以参与乙烯信号转导(Ding et al., 2019) 。
表1 1-MCP不同处理下TFs的分布
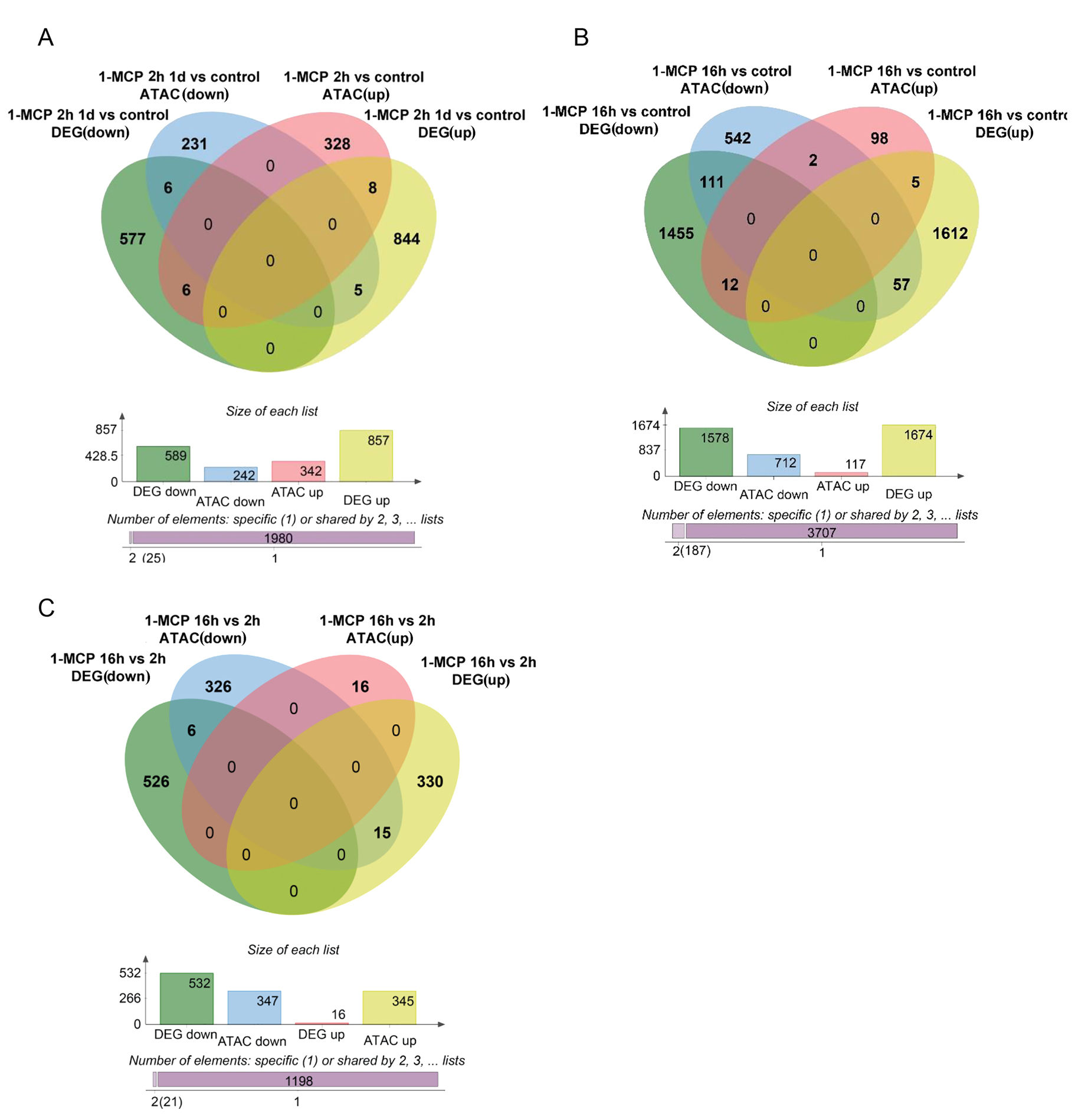
图4 1-MCP不同处理的木瓜果实样品中染色质可及性和基因表达的差异分析。Venn图显示差异表达基因(differentially expressed genes,DEGs)和DARs相关基因之间的重叠。黄色和绿色分别表示上调和下调的DEGs,粉色和蓝色分别表示上调和下调的DARs。
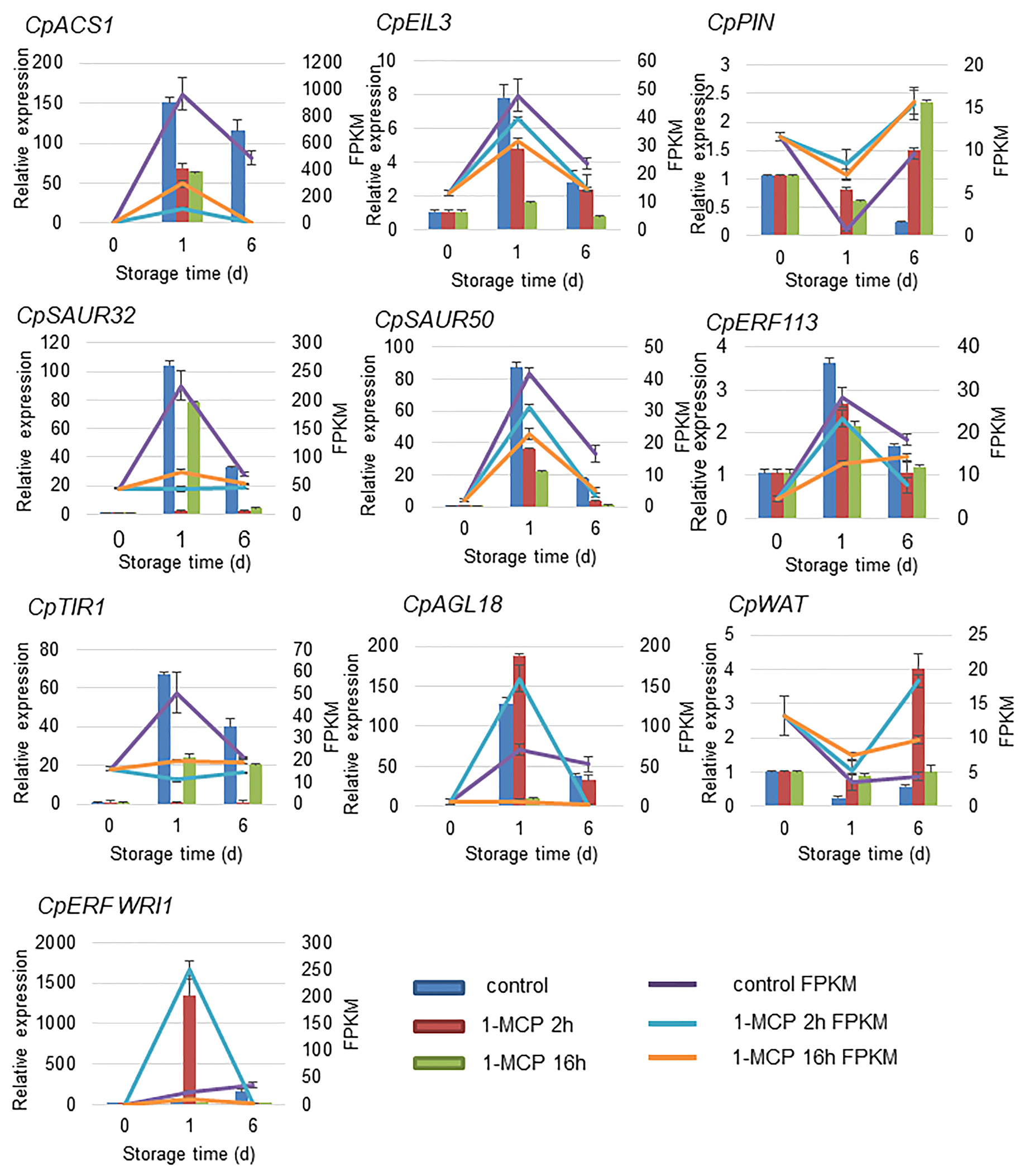
基于筛选的DEG与基因组的比对(NCBI:http://www.ncbi.nlm.nih.gov/;JGI:https://phytozome.JGI.doe.gov/pz/portal.html),与乙烯信号通路(CpEIL3、CpERF113和CpACS1)和生长素信号通路(CpWAT、CpSAUR32/50、CpPIN、CpTIR1)和CpAGL18(MADS)相关的几个基因被选择用于RT-qPCR的进一步验证。在对照组中,CpACS1、CpEIL3、CpERF113、CpSAUR32、CpSAURL50、CpTIR1和CpAGL18的表达水平在处理后1天(DAT)增加,然后在6 DAT降低(图5)。1-MCP处理显著抑制了它们的表达水平,特别是在长期处理组。生长素相关基因PIN和WAT表现出相似的表达谱,表达随成熟而下降,但1-MCP长期处理后表达增强。这些基因表达水平与RT-qPCR结果相似,进一步说明了这些表达数据的可靠性。
图5 筛选的DEGs在1-MCP不同处理下的表达验证。RT-qPCR的结果绘制成直方图,使用RNA-seq数据中的FPKM值绘制相应的折线图。
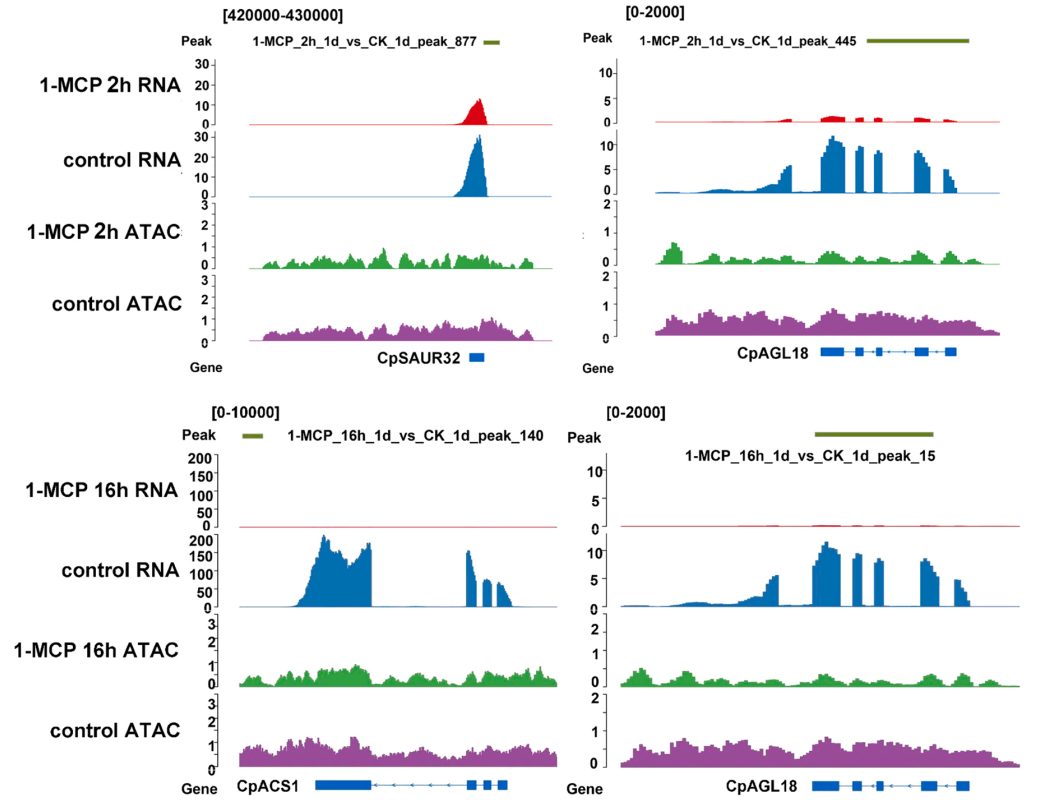
此外,整合ATAC-seq和RNA-seq的基因组可视化图谱表明,与对照相比,1-MCP短期处理抑制了CpAGL18和CpSAUR32的表达和关联染色质的开放。与对照相比,在长期1-MCP处理下,CpACS1和CpAGL18的表达和关联染色质的开放和减少,而在短期和长期1-MCP处理下,CpAGL18都受到影响(图6)。
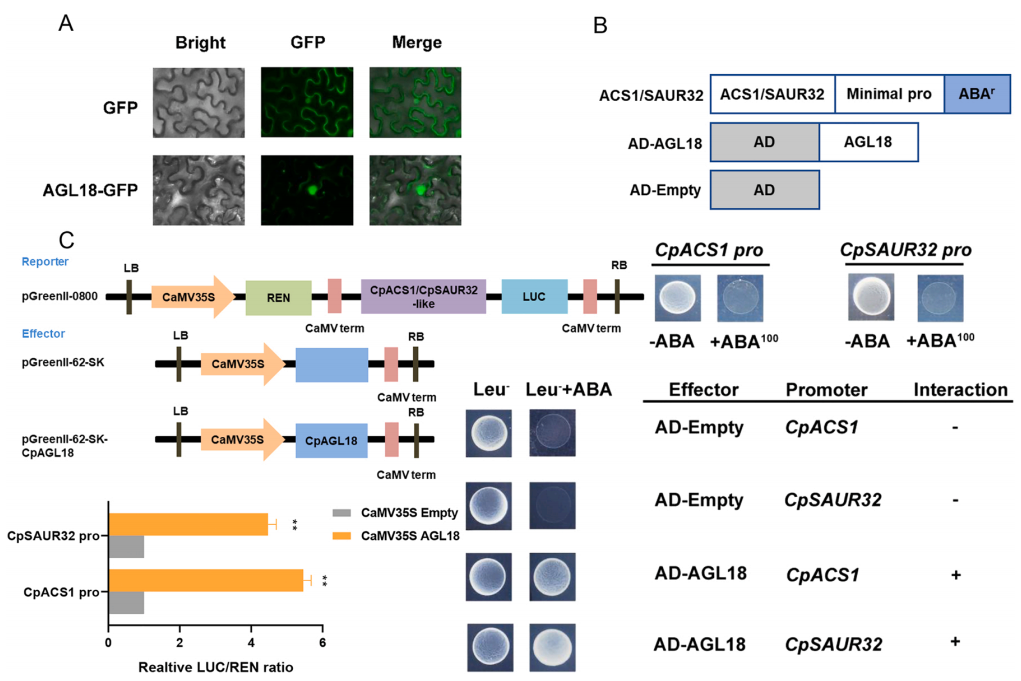
进行酵母单杂交实验(Y1H)以评估CpAGL18蛋白与CpACS1和CpSAUR32启动子区的结合活性。在酵母中未观察到CpACS1和CpSAUR32的启动子活性。用重组CpAGL18 AD转化的Y1H报告菌株和含有CpACS1和CpSAUR32启动子的酵母细胞在AbA选择培养基下生长良好,表明CpAGL18能与CpACS1和CpSAUR2启动子结合(图7B)。此外,采用双荧光素酶报告基因实验来鉴定CpACS1和CpSAUR32的启动子活性。分析表明,当CpAGL18存在时,CpACS1和CpSAUR32显示出比对照更高的LUC/REN。这些结果表明,CpAGL18显著诱导了CpACS1和CpSAUR32的表达(图7C)。
图7 检测CpAGL18与其靶基因之间的相互作用。(A)CpAGL18蛋白的亚细胞定位;(B)Y1H显示CpAGL18与CpSAUR32和CpACS1启动子相结合;(C)双荧光素酶报告基因实验中CpACS1和CpSAUR32的转录激活。
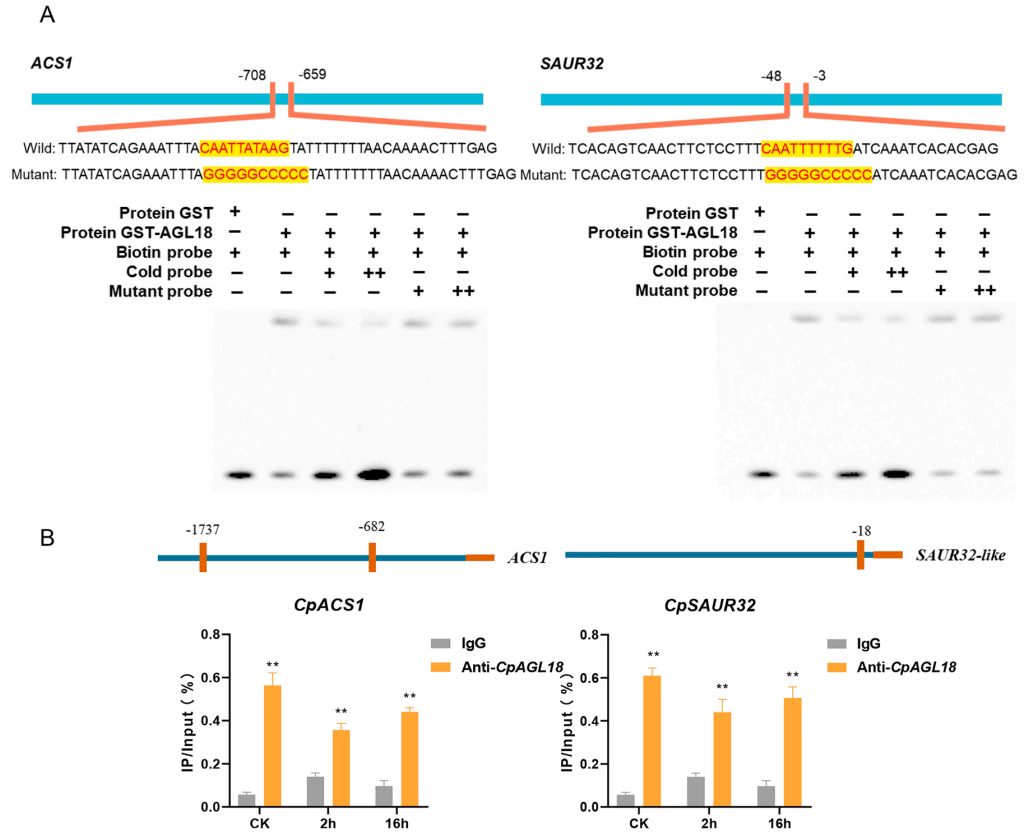
EMSA测定了CpAGL18在体外与CpACS1和CpSAUR32的启动子直接相互作用。CpAGL18与CpACS1和CpSAUR32的CarG box元件相互作用,导致迁移率的变化。增加突变片段并没有显著消除结合复合物,表明DNA-蛋白质相互作用的特异性。与非特异性抗体(IgG)相比,ChIP qPCR实验显示CpACS1和CpSAUR32启动子区的AGL18抗体显著富集(图8B)。这些结果证实了CpAGL18与CpACS1和CpSAUR32的启动子结合。
2、ATAC-seq和RNA-seq的联合分析显示CpAGL18和与生长素和乙烯相关的八个基因在果实成熟中可能具有重要作用。
3、体内和体外试验表明,CpAGL18结合并激活CpACS1和CpSAUR32的表达,从而参与乙烯和生长素信号通路,并影响木瓜果实成熟过程的调控。
文献案例二
期刊:New Phytologist
发表时间:2020.2.18
影响因子:10.323
技术手段:亚细胞定位、RT-qPCR、WB、ATAC-seq、RNA-seq、Y2H、Co-IP、BiFC
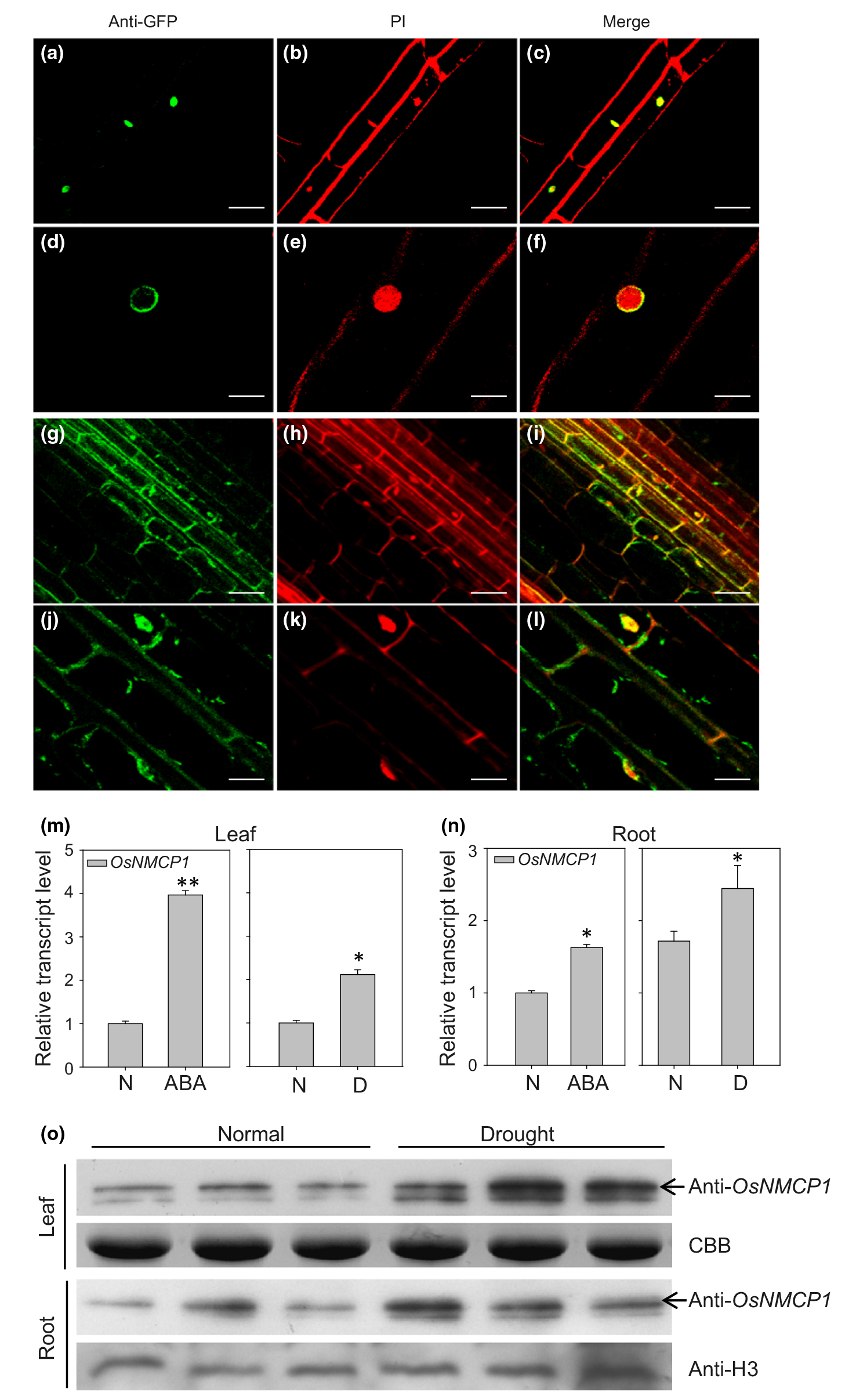
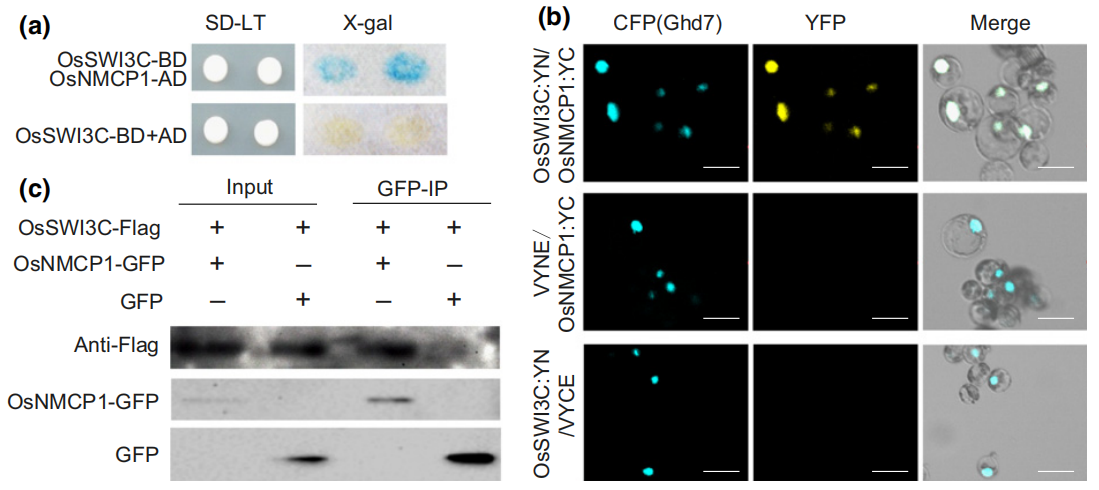
该文章的研究对象是核纤层样蛋白OsNMCP1与染色质重塑复合物OsSWI3C,通过ATAC-seq和RNA-seq联合分析OsNMCP1过表达转基因株系和OsSWI3C干扰转基因株系在干旱和正常条件下染色质的可及性和下游基因的表达情况,从而揭示OsNMCP1和OsSWI3C调控干旱和根生长相关基因中的作用。这篇文章中其他实验都相对完整,但ATAC-seq和RNA-seq联合分析让文章提升了一个层次,非常值得想发高分文章的老师学习哦!想看组学实验结果可以直接跳到结果4哦!
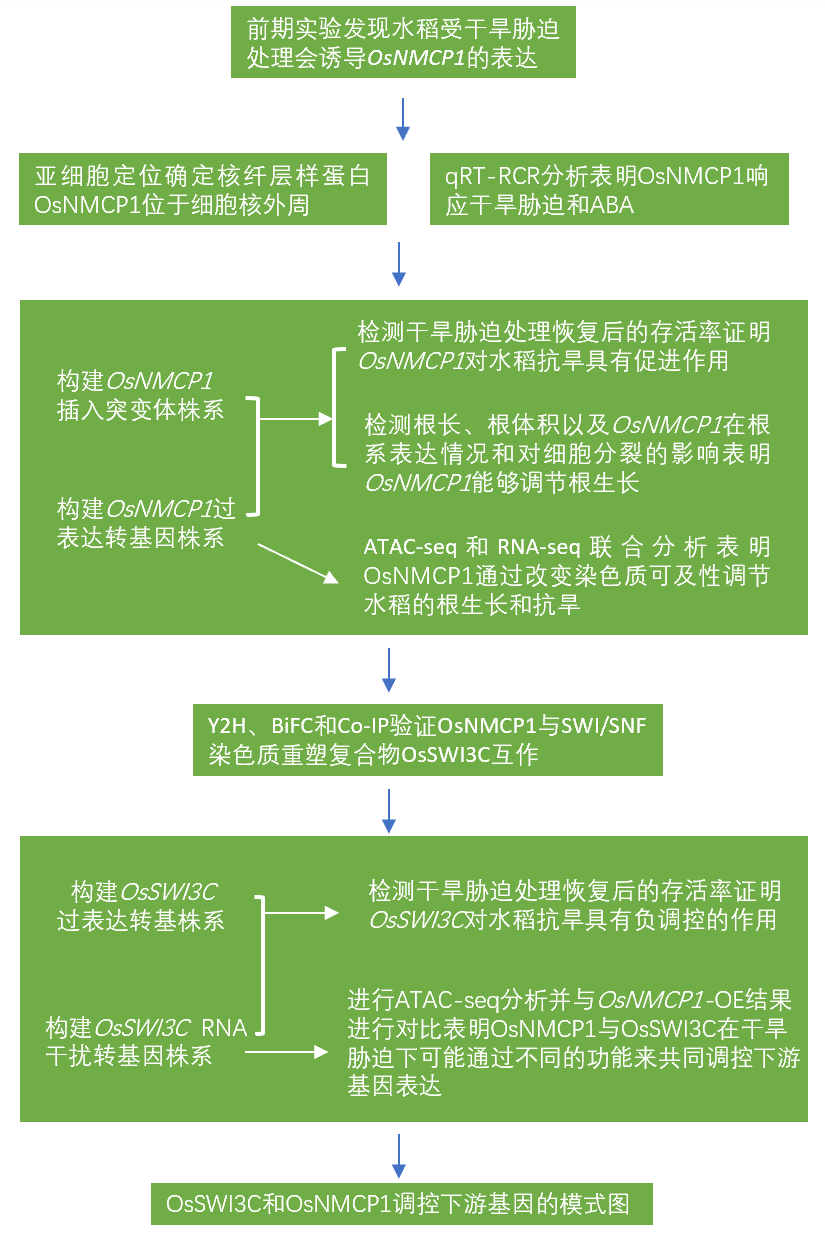
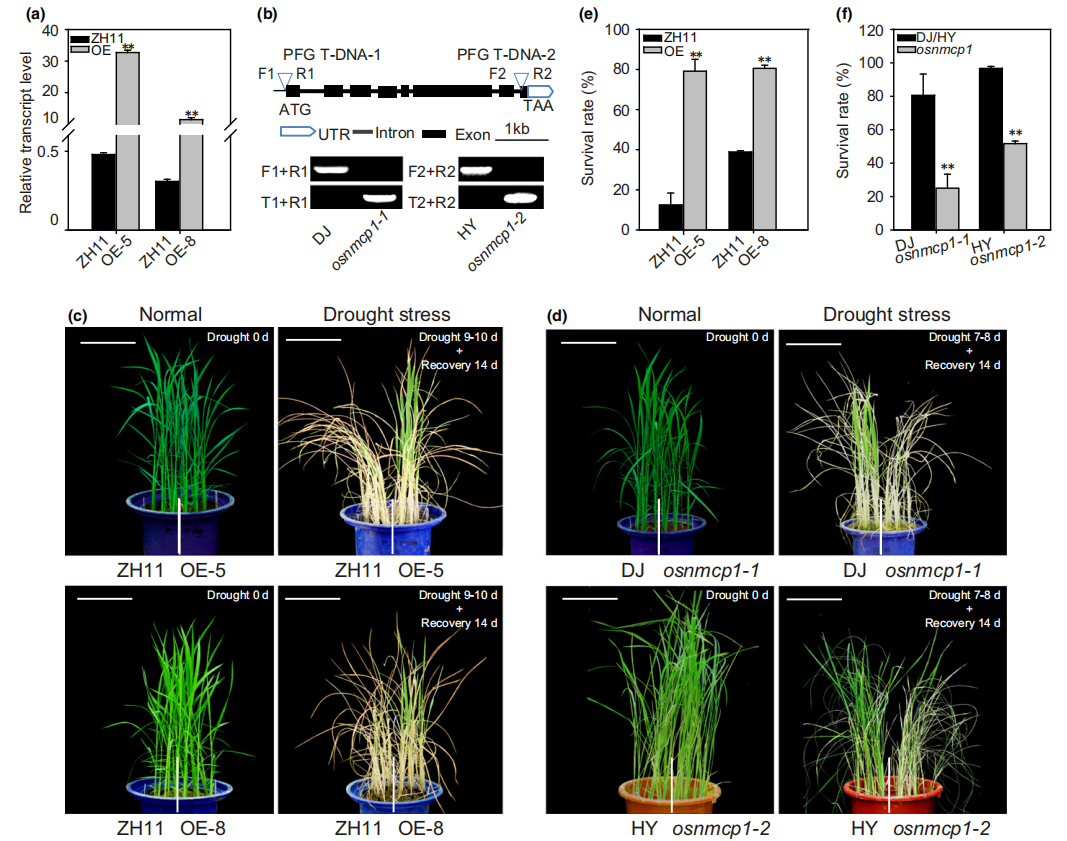
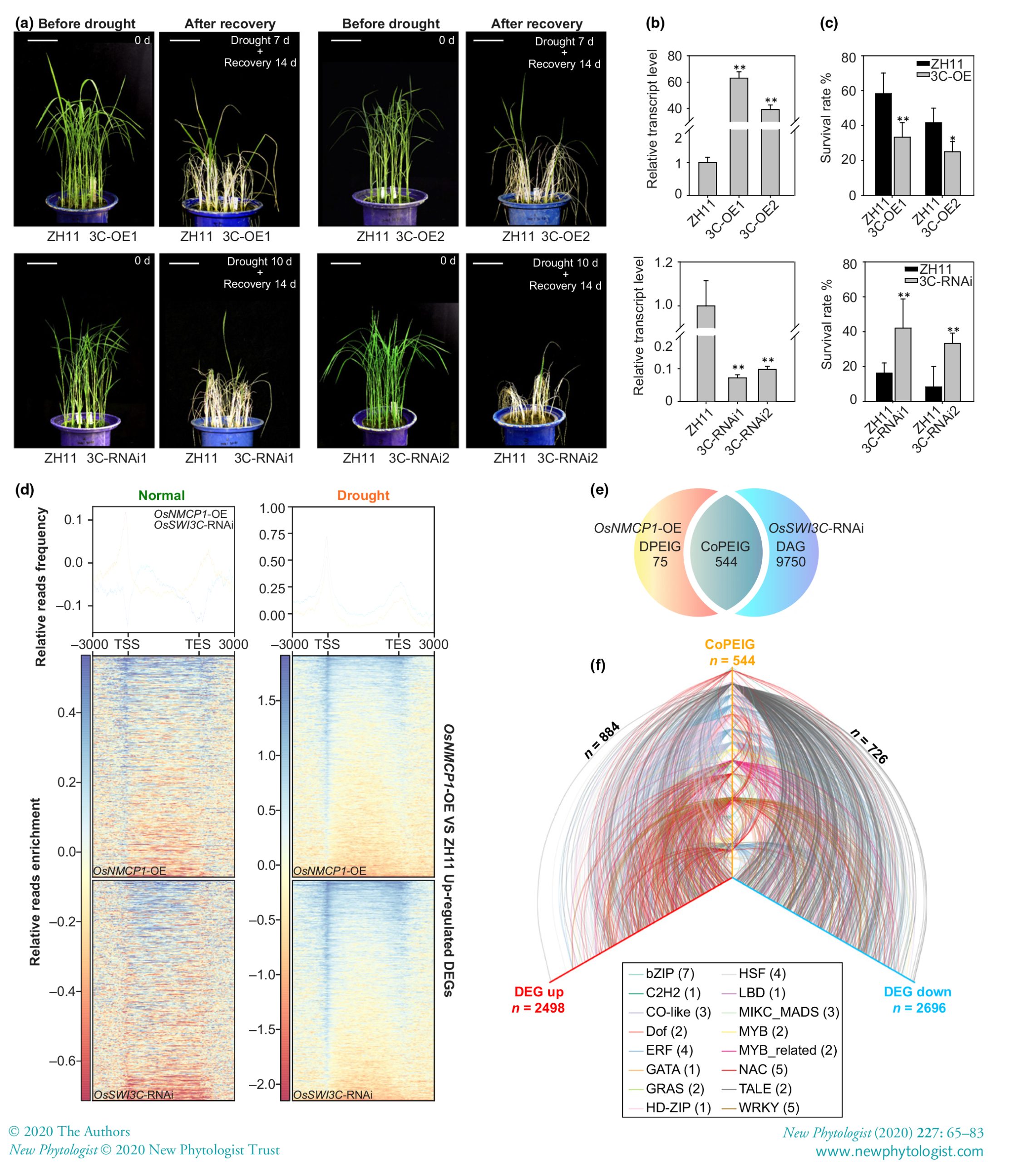
图10水稻苗期干旱胁迫处理下osnmcp1突变体植物和OsNMCP1–OE植物的表型。(a)OsNMCP1在ZH11(Oryza sativa ssp.japonica)和两个独立系OsNMCP1–OE中的qRT-PCR表达分析;(b)OsNMCP1基因结构示意图, osnmcp1-1和osnmcp1-2突变体的T-DNA插入和基因分型。ATG,起始密码子;TAA,终止密码子;PFG,T-DNA插入载体;F和R、正向和反向基因组引物;(c)ZH11和OsNMCP1–OE植物在正常生长条件和苗期干旱胁迫处理下的表现;(d)野生型水稻Dongjing(DJ)或Huayoung(HY)(O.sativa ssp.japonica)以及osnmcp1-1和osnmcp1-2突变体系在正常生长条件和苗期干旱胁迫处理下的表现;(e)ZH11和OsNMCP1–OE植物在干旱胁迫处理补水后的存活率比较;(f)干旱胁迫处理补水后DJ/HY和osnmcp1突变体植物的存活率比较。误差棒表示三个重复的SD值。Bar,10cm。统计显著性由 T检验确定:**,P<0.01。
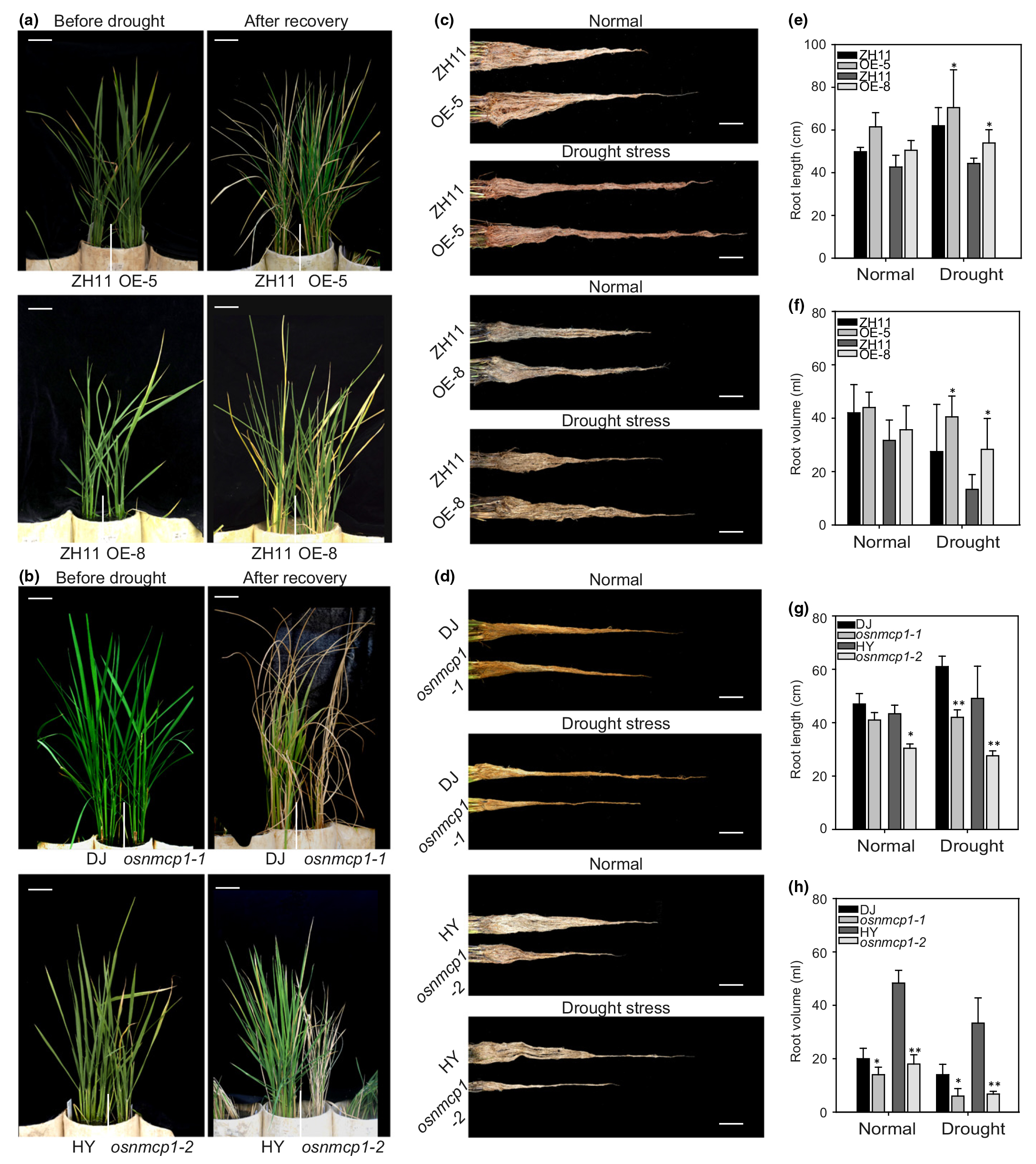
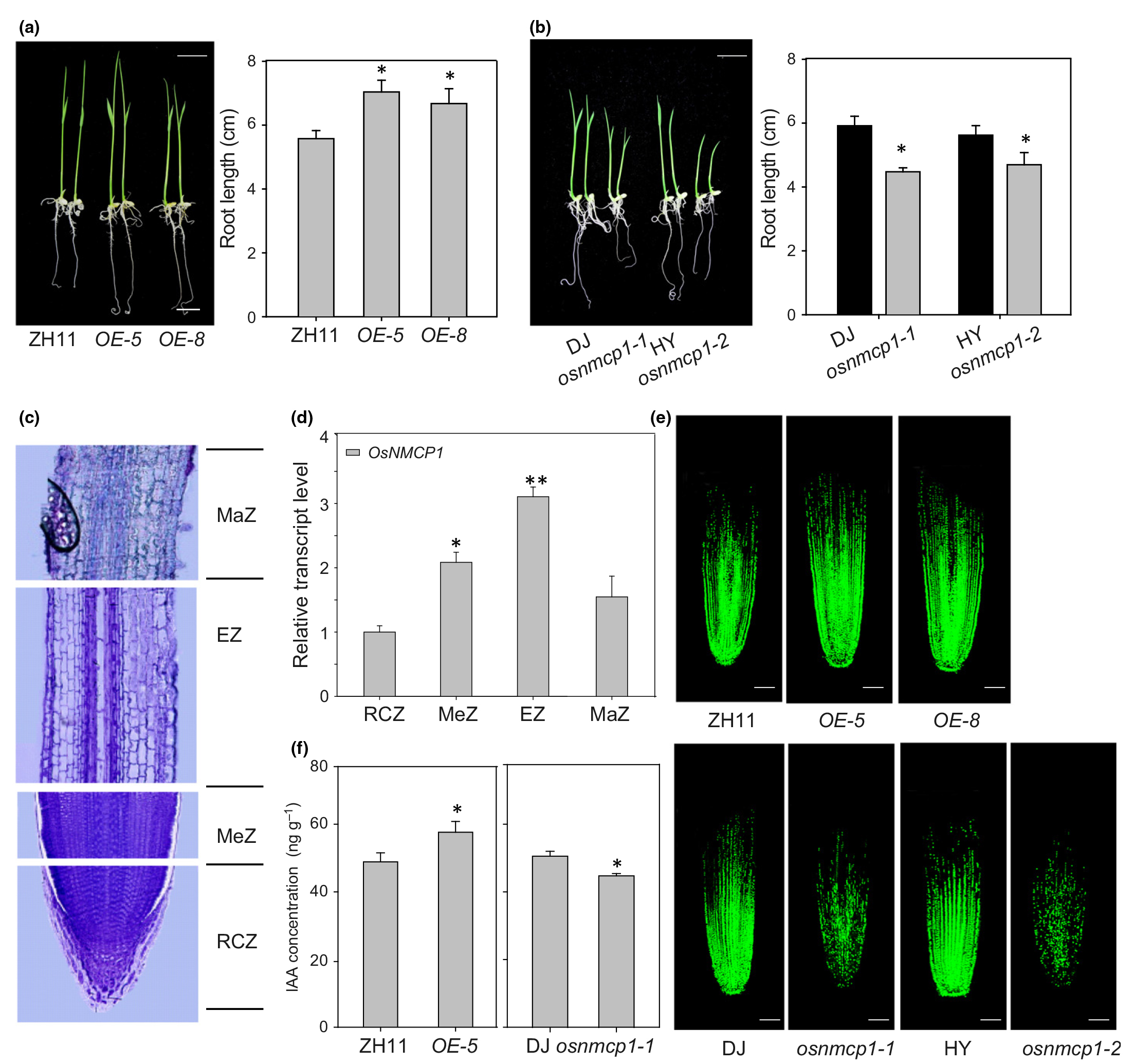
由于没有观察到野生型和转基因植物离体叶片的失水率存在显著差异,因此研究人员检测了正常和干旱胁迫条件下种子成熟阶段植株的根的最大根长和根体积。OsNMCP1–OE在正常生长条件下比ZH11具有更长的根长和更大的根体积,并且在干旱胁迫条件下差异更显著(图11c,e,f)。在正常和干旱胁迫条件下,与DJ/HY相比,osnmcp1突变体显示出明显更短的根长和更小的根体积(图11d,g,h)。这些结果共同表明,OsNMCP1在调节抗旱性方面发挥了积极作用,这可能部分归因于根系生长的增加。
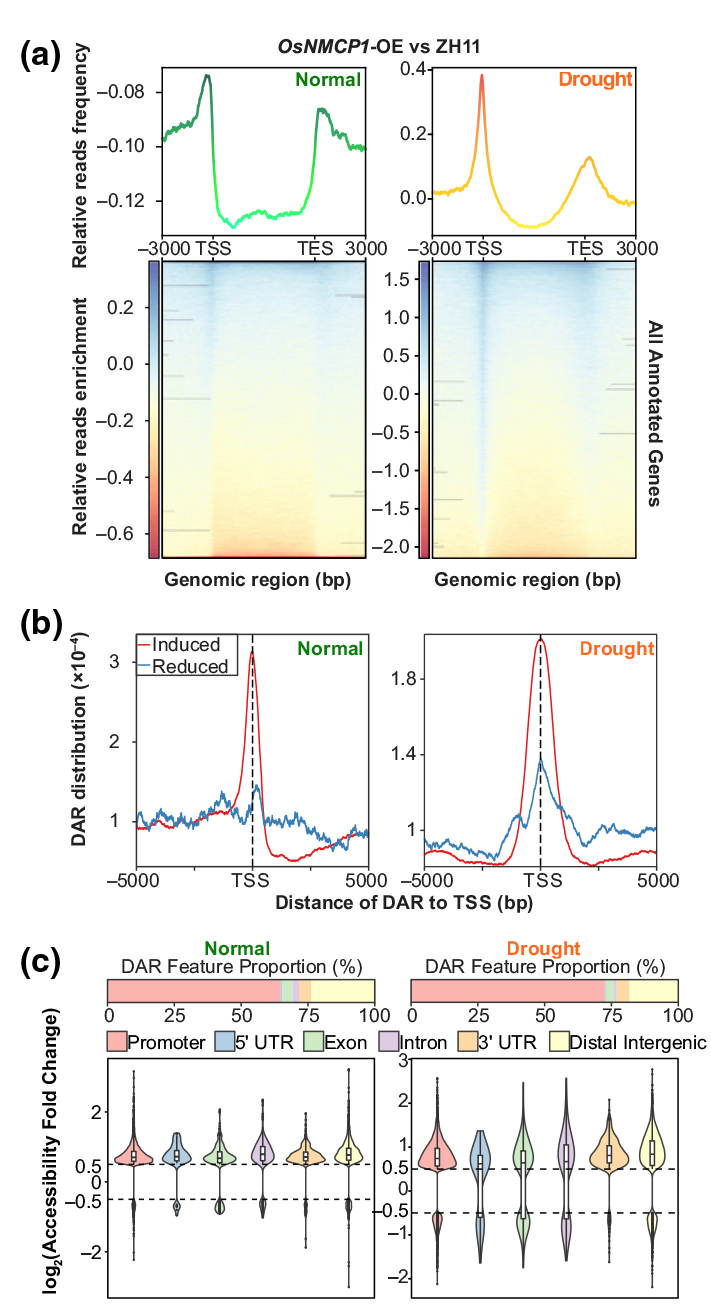
此外,上调DARs的基因组分布特征呈现出明显的启动子富集模式,其中>65%的peak在启动子区域(TSS的-3500至+100 bp区域)(图13c)。这些结果表明OsNMCP1可能主要作为染色质可及性的正调节蛋白,并通过提高其启动子区的可及性来调节下游基因的表达。
图13 OsNMCP1过表达改变了水稻染色质的可及性。(a)在正常和干旱条件下,OsNMCP1-OE与ZH11的ATAC-seq相对覆盖率曲线。上面的线图指注释基因的基因组区域的OsNMCP1-OE标准化reads的相对分布频率。下面的热图表明注释基因的-3000bp至+3000bp基因组区域中的相对reads覆盖率;(b)peak曲线图显示了与ZH11相比OsNMCP1-OE上调或下调DARs在所有注释基因的基因组区域的分布,红色表示上调DARs,蓝色表示下调DARs;(c)DARs的基因组区域分布比例。上面条形图显示了OsNMCP1-OE上调DARs的基因组分布区域的比例(仅考虑DARs最近基因的特征)。下面小提琴图中的log2(Accessibility Fold Change)说明了每个基因组分布区域中DARs的丰度。
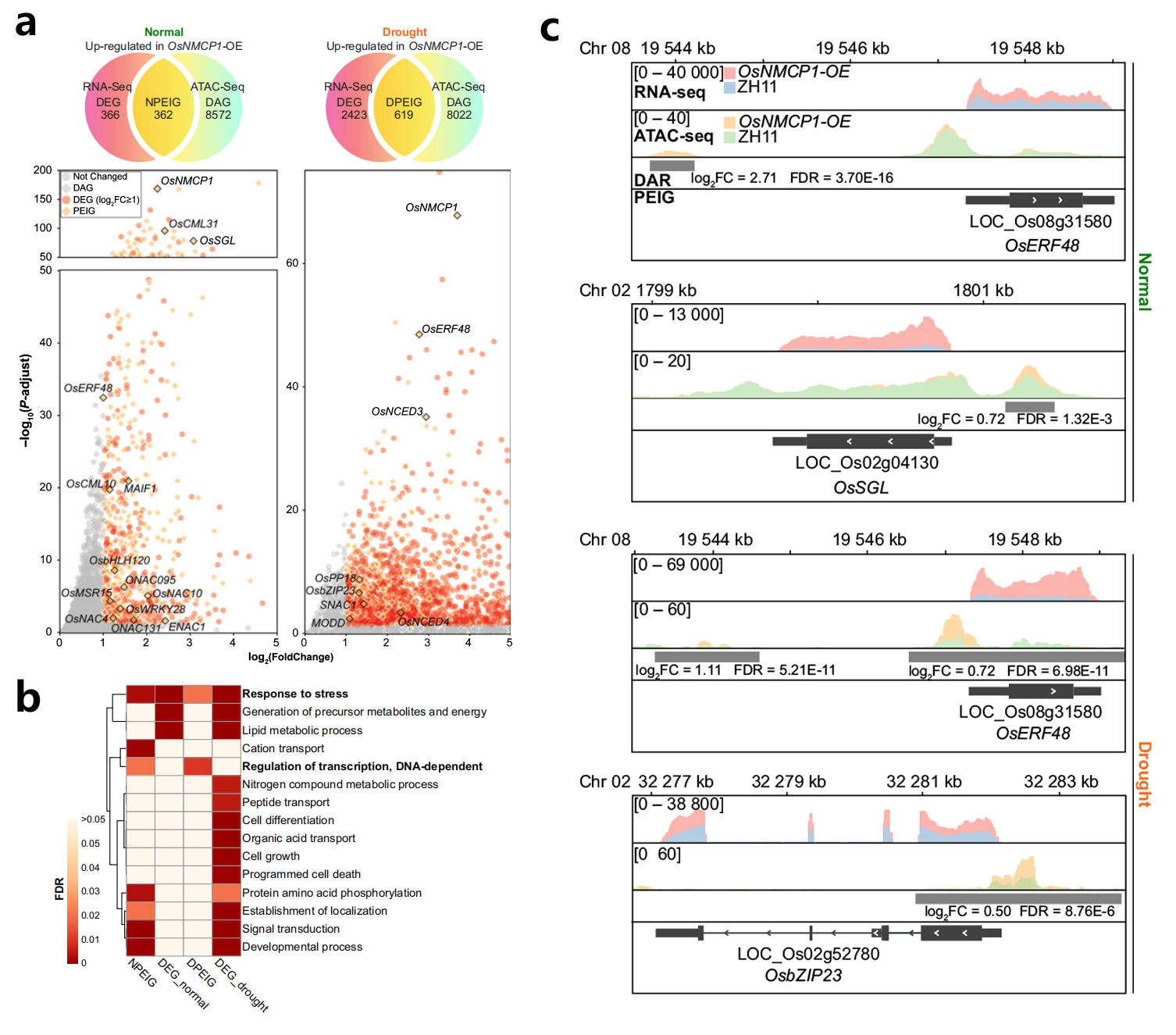
为了证实这一假设,对用于ATAC-seq的相同植物样本进行了RNA-seq分析,以评估染色质的这些变化是否与相邻基因表达的变化有关。整合分析在正常和干旱条件下DAGs与上调DEGs的重叠部分,分别发现362和619个相同基因,占上调DEGs总数的49.93%和20.35%(图14a)。研究人员将这些基因定义为正常(N)和干旱(D)条件下的启动子可及性和表达增加基因(Promoter-accessibility and expression increased genes,PEIGs),即NPEIGs和DPEIGs(图14a)。对PEIGs和DEGs的GO富集分析表明,这些基因都与应激反应功能相关(图14b),这与OsNMCP1在抗旱性中的作用有关。
值得注意的是,两个PEIGs数据集,都在“转录调节”GO term中富集。而DEGs不在“转录调节”GO term中富集。进一步研究了PEIGs的组成,发现17.4%的NPEIG和7.9%的DPEIG都编码转录因子(TF),这是除了未鉴定的蛋白质之外的最大类别,包括SNAC1和OsbZIP23(图14a,c)。这些结果表明OsNMCP1可能调节与干旱反应和根系生长相关的基因。
据报道,CRC通过改变染色质可及性参与转录调节。因为OsSWI3C–RNAi植物表现出更强的抗旱性,研究人员进一步研究了OsSWI3C的抑制是否影响OsNMCP1–OE中基因的染色质可及性。将OsSWI3C-RNAi的ATAC-seq结果与OsNMCP1–OE的ATAC-seq结果进行对比,在干旱胁迫下OsNMCP1–OE的上调基因与OsSWI3C-RNAi高度相似,在TSS区域强烈富集(图16d)。但是正常条件下生长的样本,OsSWI3C-RNAi株系在TSS区域分布显著性降低(图16d)。对比OsSWI3C-RNAi和OsNMCP1-OE株系的结果,显示它们有544个相同基因(CoPEIGs),占DPEIGs的87.88%,表明OsNMCP1和OsSWI3C的共调控(图16e)。从水稻表达数据库中提取与这些TFs相关系数大于0.7的RNA-seq的数据进行分析,结果显示有884个和726个共表达相关基因(图16f)。这些结果表明OsNMCP1与OsSWI3C在干旱胁迫下可能通过不同的途径来共同调控下游基因表达。
2、OsSWI3C在OsNMCP1介导的基因表达调控中发挥重要作用。
3、调控模式:
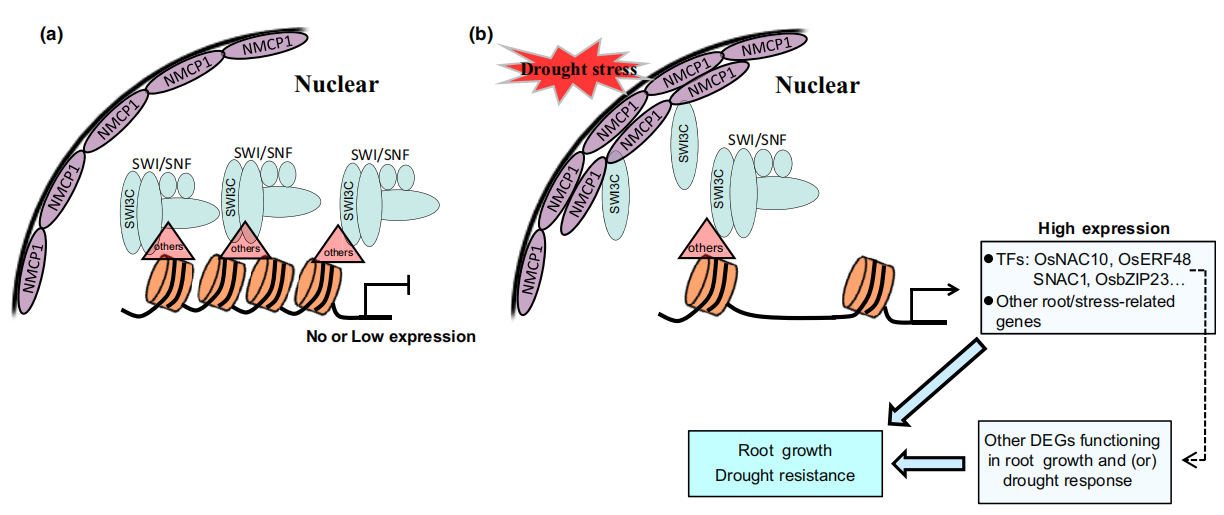
SWI/SNF染色质重塑复合物亚基OsSWI3C被招募来改变核小体的结构,抑制染色质的开放,导致与根生长和抗旱相关基因的沉默。干旱胁迫诱导OsNMCP1蛋白表达,OsNMCP1蛋白水平的升高可能导致更多的核纤层样核外周蛋白OsNMCP1与OsSWI3C“相遇”,并与OsSWI3C相互作用。这种相互作用可能会使SWI/SNF复合物释放OsSWI3C分子,从而增加了与根生长和抗旱相关基因中的染色质开放程度,从而导致这些基因表达上调。
Cai J, Wu Z, Song Z, et al. 2022. ATAC-seq and RNA-seq reveal the role of AGL18 in regulating fruit ripening via ethylene-auxin crosstalk in papaya. Postharvest Biology and Technology 191.
Petrasek J, Friml J. 2009. Auxin transport routes in plant development. Development 136:2675-2688.
Sivakumar D, Wall MM. 2013. Papaya Fruit Quality Management during the Postharvest Supply Chain. Food Reviews International 29:24-48.
Tian Y, Lu Xy. 2006. The molecular mechanism of ethylene signal transduction. South African Journal of Botany 72:487-491.
Stuurman N, Heins S, Aebi U. 1998. Nuclear lamins: their structure, assembly, and interactions. Journal of Structural Biology 122: 42–66.
Dittmer TA, Misteli T. 2011. The lamin protein family. Genome Biology 12: 222.
Yang J, Chang Y, Qin Y, et al. 2020. A lamin-like protein OsNMCP1 regulates drought resistance and root growth through chromatin accessibility modulation by interacting with a chromatin remodeller OsSWI3C in rice. New Phytol 227:65-83.
Sarnowski TJ, Swiezewski S, Pawlikowska K, Kaczanowski S, Jerzmanowski A. 2002. AtSWI3B, an Arabidopsis homolog of SWI3, a core subunit of yeast Swi/Snfchromatin remodeling complex, interacts with FCA, a regulator of flowering time. Nucleic Acids Research 30: 3412–3421.
Clapier CR, Cairns BR. 2009. The biology of chromatin remodeling complexes.
Annual Review of Biochemistry 78: 273–304.