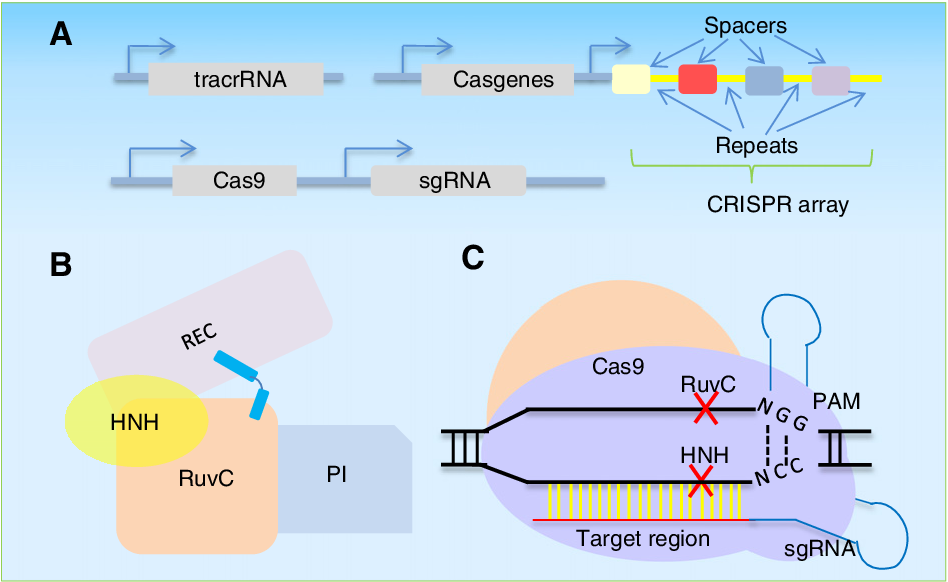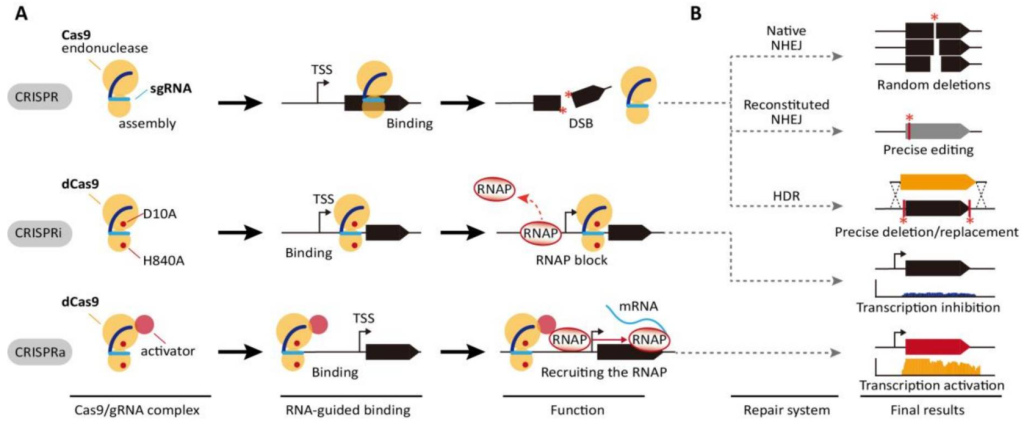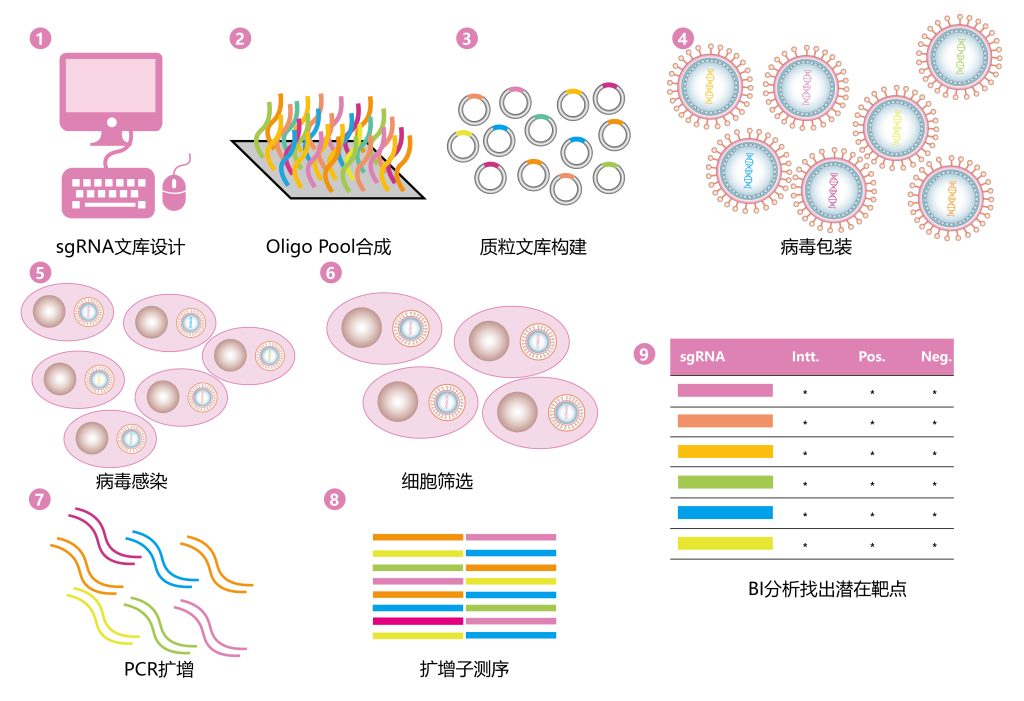
图2 sgRNA文库构建流程(图片来源:伯远生物)。
CRlSPR/Cas9 gRNA文库可分为以下三种:
1)基因敲除文库(CRISPR KO):利用野生型Cas9或Cas9缺刻酶(Cas9 nickase,nCas9)切割DNA,当细胞自身发生随机修复时,可在靶位点(通常是编码区域)引入随机突变,主要用于编码基因的功能筛选。
2)基因激活文库(CRISPRa):转录激活功能域融合无催化活性的Cas9(dCas9),以激活靶位点(通常是启动子区和其他非编码调控区)调节基因表达,主要用于编码基因的功能获得筛选或非编码区域的调控功能筛选。
3)基因抑制/沉默文库(CRISPRi):转录阻遏物融合dCas9,以抑制靶位点(通常是启动子区和其他非编码调控区)调节基因表达,编码基因的功能缺失筛选或非编码区域的调控功能筛选。
3、可用于全基因组范围筛选,有利于新靶点发现;
4、编辑效果假阳性比率较低,靶点命中率高;
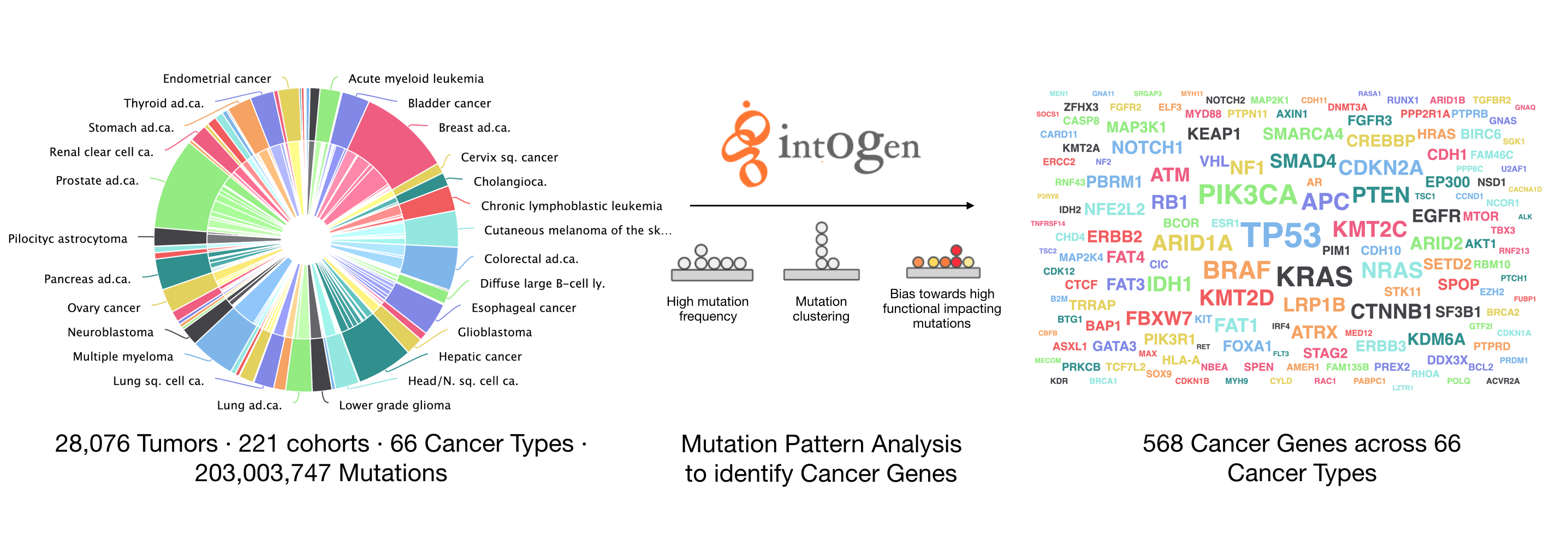
图4 肿瘤驱动基因与肿瘤(图片源自:IRB Barcelona)。
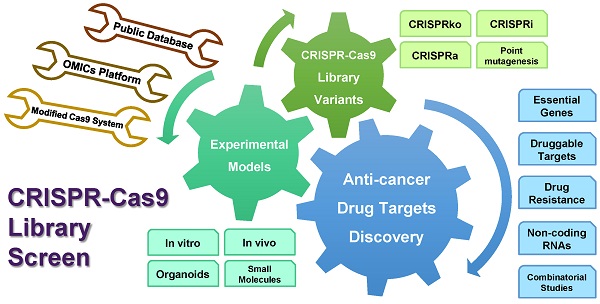
介于CRlSPR/Cas9 gRNA文库可同时靶向全基因组不同基因,成为实现高通量基因失活的绝佳工具。2022年4月11日发表在Theranostics杂志上的“CRISPR-Cas9 library screening approach for anti-cancer drug discovery: overview and perspectives”一文介绍了近年来兴起的高通量CRISPR-Cas9 gRNA文库基因组筛选技术及其工作原理,并着重介绍了其在抗癌药物发现中的各种应用的优势,包括功能和靶点识别、非编码RNA信息、小分子的作用以及药物靶点的发现(图5)。
图5 用于抗癌药物发现的CRISPR/Cas9 sgRNA文库筛选(Chan et al., 2022)。
2、筛选合成致死作用靶点,实现精准治疗
在描述CRISPR/Cas9 sgRNA文库应用之前,首先让我们先来了解下容易混淆的几大肿瘤相关基因的分类吧!
(1)致癌基因(Oncogene):也称为癌基因,是一类能够促使正常细胞转化为癌细胞的基因,与恶性肿瘤发生有关。常见的致癌基因家族有Ras家族、MYC家族、Src家族、Sis家族等;
(2)原癌基因(Proto-oncogene):一类存在于正常细胞基因组中的基因,与细胞的增殖、生长、分化相关,功能正常的原癌基因并不会导致癌症,当其发生片段丢失、点突变、复制等突变后,就可能转化为致癌基因;
(3)抑癌基因(Antioncogene):又称肿瘤抑制基因、抗癌基因,是1980年代发现的一类抑制细胞过度生长、增殖,从而遏制肿瘤形成的基因。迄今已从细胞中分离鉴定出百余种抑癌基因,如常见的Rb1(人类第一个被发现的抑癌基因)、p53、p16、APC、NM23等。
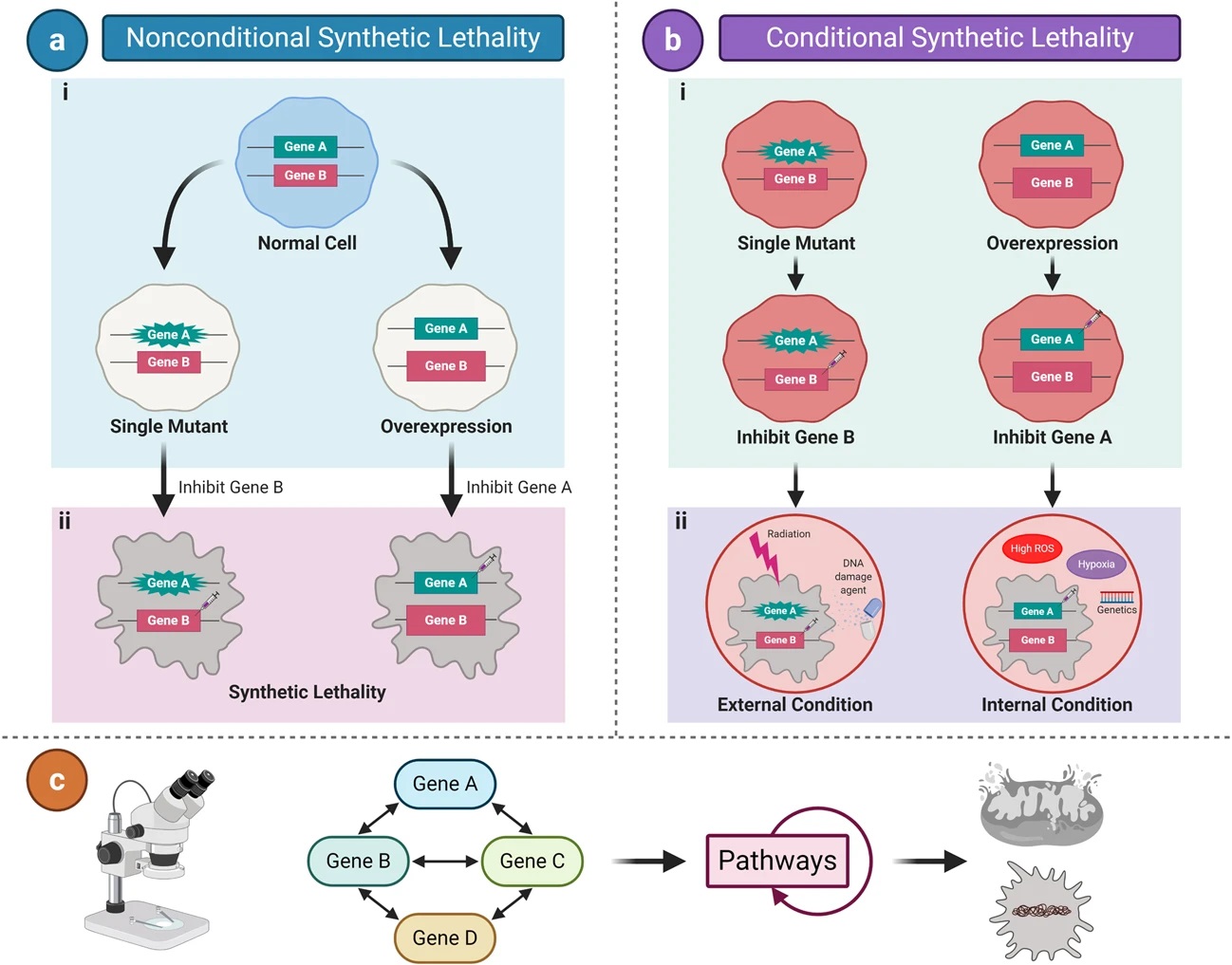
近年来,合成致死(Synthetic lethality,SL)即两个非致死基因同时失活导致细胞死亡的现象,作为肿瘤学疗法开发的新兴领域,基于该机理的新药进展引发了业界瞩目,SL通常分为两大类:非条件/原始SL和条件SL(图6)。
图6 合成致死的分类(Li et al., 2020)。
那么如何利用CRISPR/Cas9 sgRNA文库寻找更多具有合成致死作用的基因对呢?目前一般有两种逻辑:(1)直接设计双重打靶的gRNA载体,即在一个细胞内同时靶向两个基因,筛选具有合成致死作用的基因对(Shen et al., 2017);(2)在单个抑癌基因敲除的细胞系上进行文库筛选,筛选出与敲除的抑癌基因具有合成致死作用的靶点(Feng et al., 2022)。
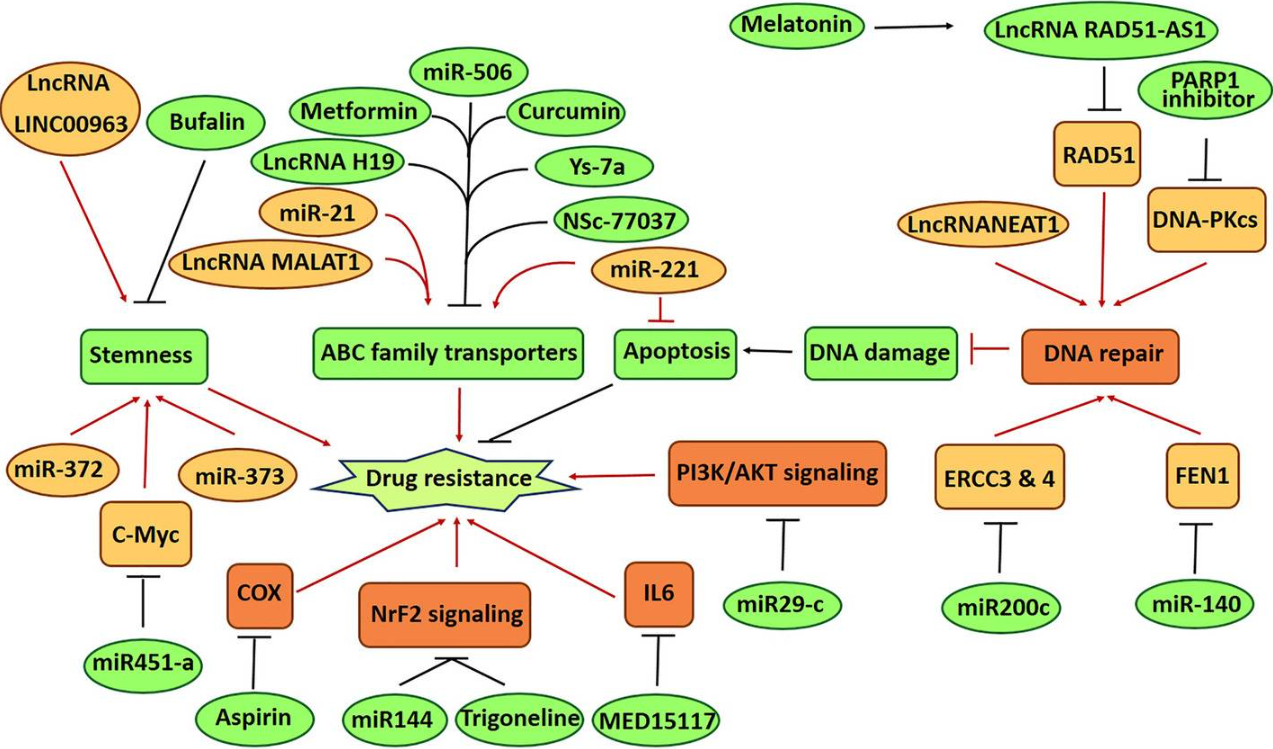
3、寻找肿瘤耐药相关基因
众所周知,肿瘤耐药是导致肿瘤治疗失败的主要原因之一,极大地限制了癌症药物的选择和使用,因此探讨肿瘤的耐药机制具有重要临床意义(图7)。耐药可分为内源性(或从头)耐药(肿瘤细胞在药物治疗前就存在耐药因子)或获得性耐药(在药物治疗过程中产生耐药性),并且,耐药的一个重要特征是对一种药物的耐药可能导致对其他药物的耐药,这就是多药耐药(Multidrug resistance,MDR)。
Chen C, Sha H, Yang B, et al. Establishment and optimization of genome-wide CRISPR/Cas9-sgRNA screening system in THP1 cell line for functional oncogenes and tumor suppressor genes[J]. Scientia Sinica(Vitae), 2016.
Cho S, Shin J, Cho BK. Applications of CRISPR/Cas System to Bacterial Metabolic Engineering. Int J Mol Sci. 2018;19(4):1089.
Feng X, Tang M, Dede M, et al. Genome-wide CRISPR screens using isogenic cells reveal vulnerabilities conferred by loss of tumor suppressors. Sci Adv. 2022;8(19):eabm6638.
Li S, Topatana W, Juengpanich S, et al. Development of synthetic lethality in cancer: molecular and cellular classification. Signal Transduct Target Ther. 2020;5(1):241.
Shen JP, Zhao D, Sasik R, et al. Combinatorial CRISPR-Cas9 screens for de novo mapping of genetic interactions. Nat Methods. 2017;14(6):573-576.
Song G, Jia M, Chen K, et al. ScienceDirect CRISPR / Cas 9: A powerful tool for crop genome editing. The Crop Journal. 2016.
Vaghari-Tabari M, Hassanpour P, Sadeghsoltani F, et al. CRISPR/Cas9 gene editing: a new approach for overcoming drug resistance in cancer. Cell Mol Biol Lett. 2022;27(1):49.