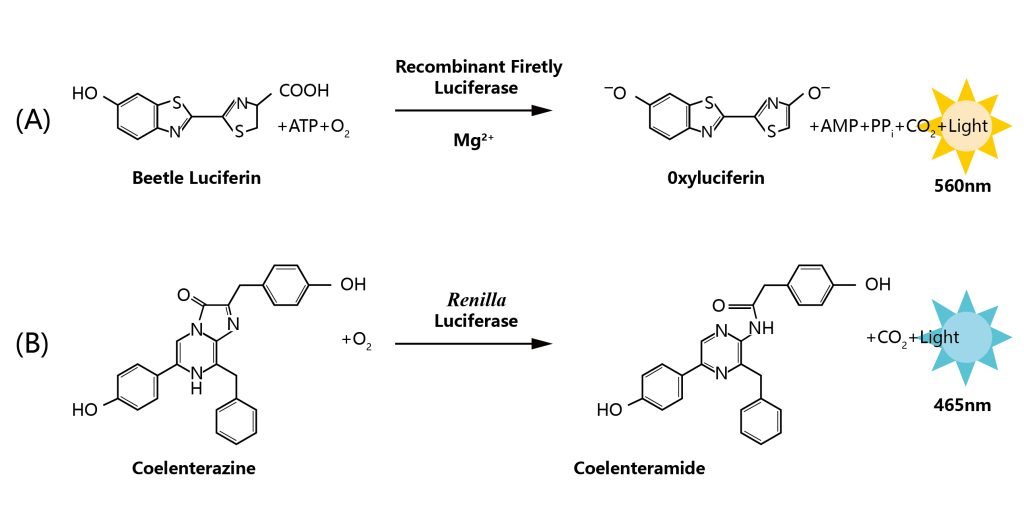
荧光素酶是生物体内催化荧光素(luciferin)或脂肪醛(firefly aldehyde)氧化发光的一类酶的总称,来自于自然界能够发光的生物。自然界存在的荧光素酶多来自萤火虫、发光细菌、发光海星、发光节虫、发光鱼、发光甲虫等。
目前,以北美萤火虫(Photinus pyralis)来源的荧光素酶基因应用的最为广泛,该基因可编码550个氨基酸的荧光素酶蛋白,是一个62kDa的单体酶,无需表达后修饰,直接具有可被检测的酶活(张菊梅等, 2001);另外,提取自海洋腔肠动物海肾(Renilla reniformis)的荧光素酶也是一种可以催化荧光素发生荧光反应的单亚基特异活性蛋白,其分子量为36kDa。同萤火虫荧光素酶一样,该蛋白质在完成转录翻译后即具有催化活性(赵斯斯, 2012)。
双荧光素酶报告基因检测的实验步骤可大致分为以下5步:
(1)构建相应的载体;
(2)将载体转染至细胞内进行表达;
(3)裂解液裂解细胞;
(4)荧光检测;
(5)数据分析得到实验结论。
双荧光素酶报告基因检测常用的载体有两种策略。第一种是两种荧光素分别位于两个载体上,即将带有海肾荧光素酶基因的质粒与报告基因质粒共转染细胞;第二种是两种荧光素酶位于同一个载体上,两种荧光素酶分别用不同的启动子启动其表达。
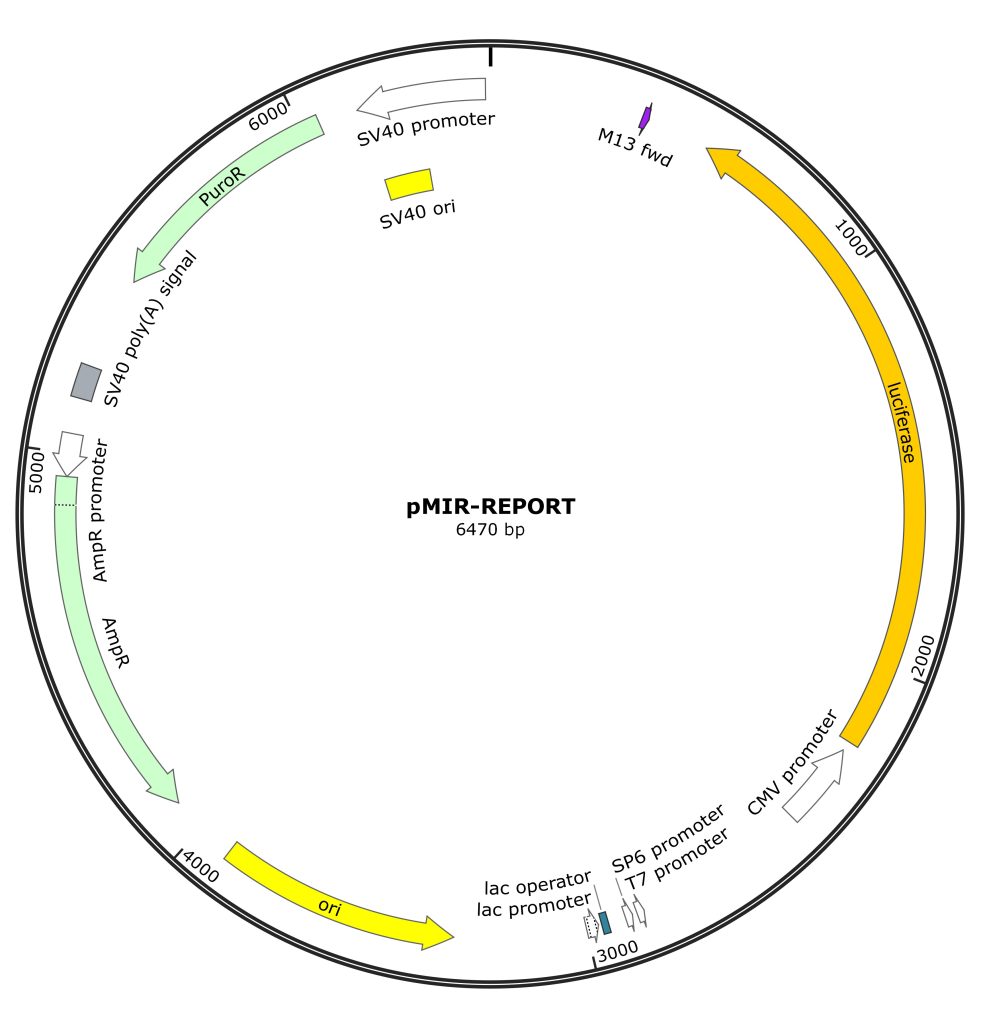
(1)pMIR-REPORT载体
pMIR-REPORT载体是美国Ambion公司开发的由CMV启动子启动,用来克隆插入miRNA靶序列,评估细胞内miRNA功能的载体。其也可用来检测siRNA的靶位点和分析3’端序列对基因表达的影响。在F-Luc的3’端包含一个多克隆位点,用于插入miRNA的结合靶序列或其他核苷酸序列。
图2 pMIR-REPORT荧光素酶报告基因空载图谱。
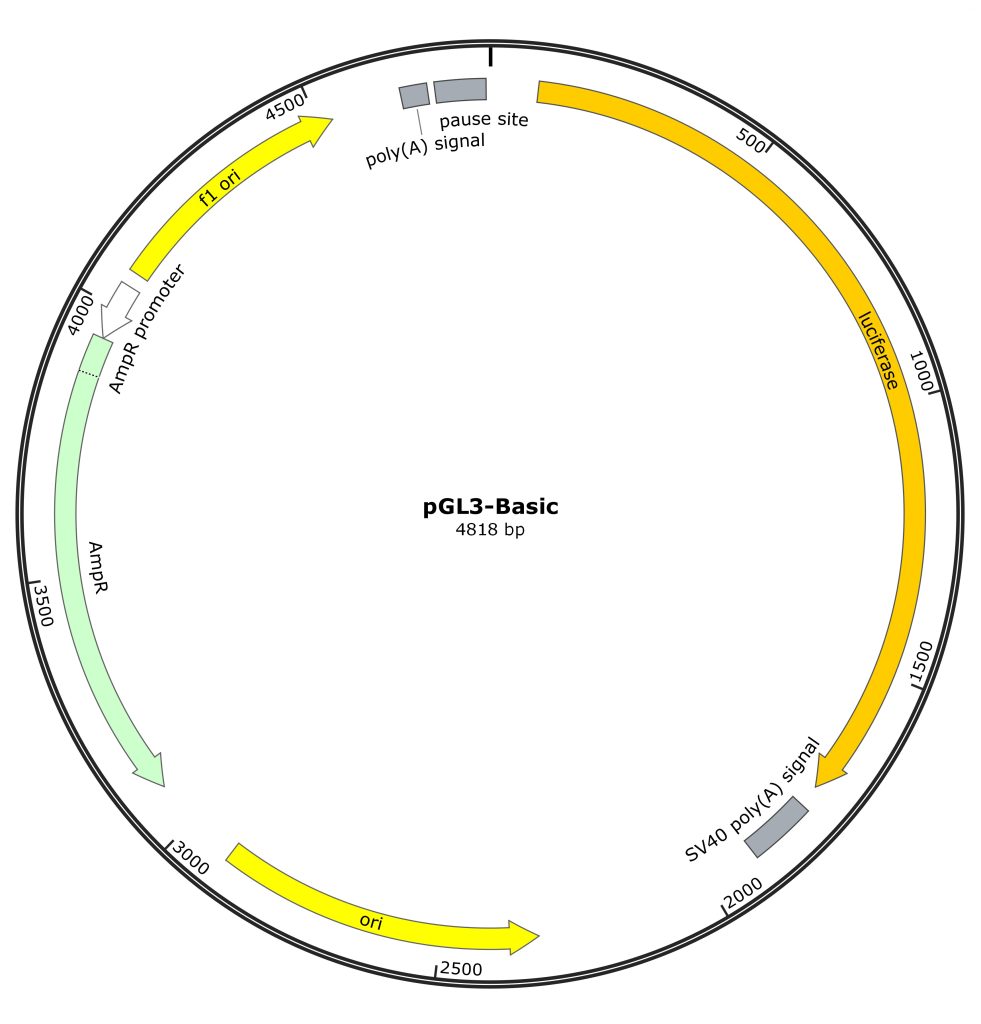
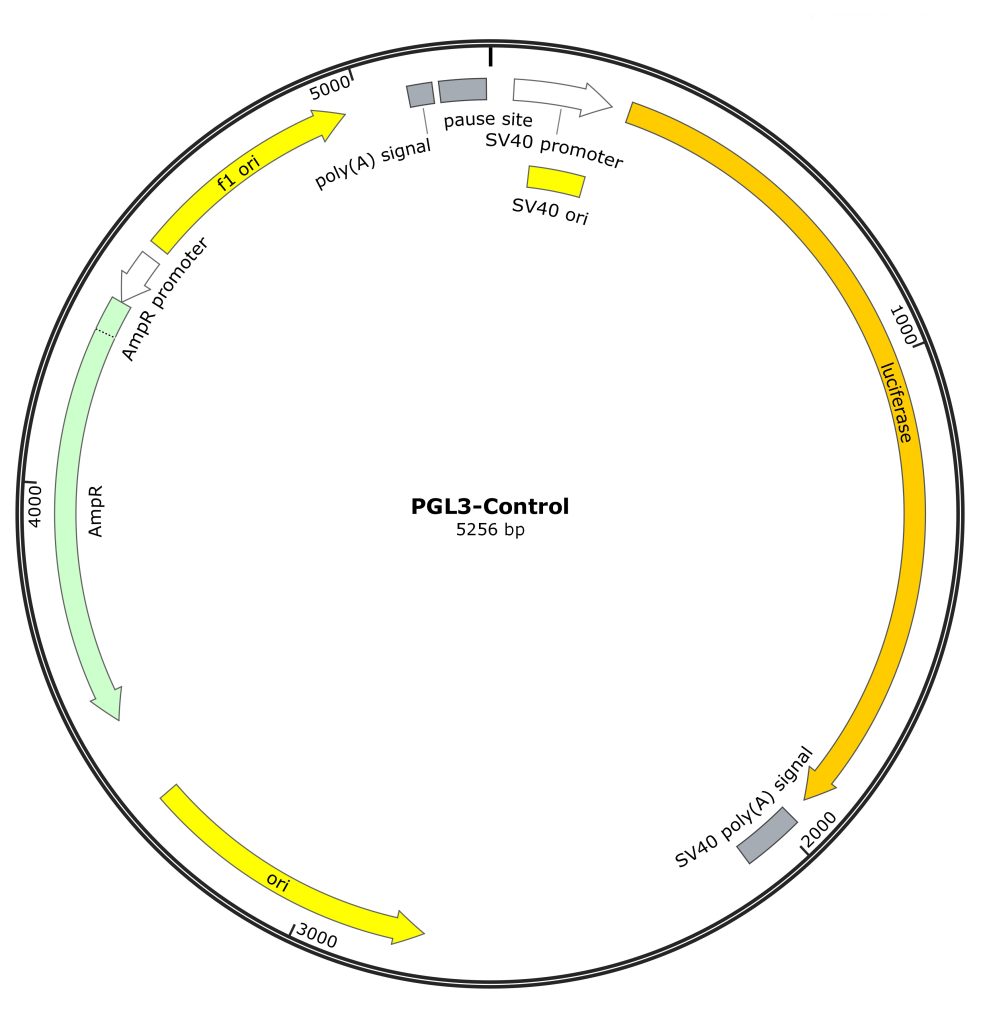
(2)pGL3系列载体
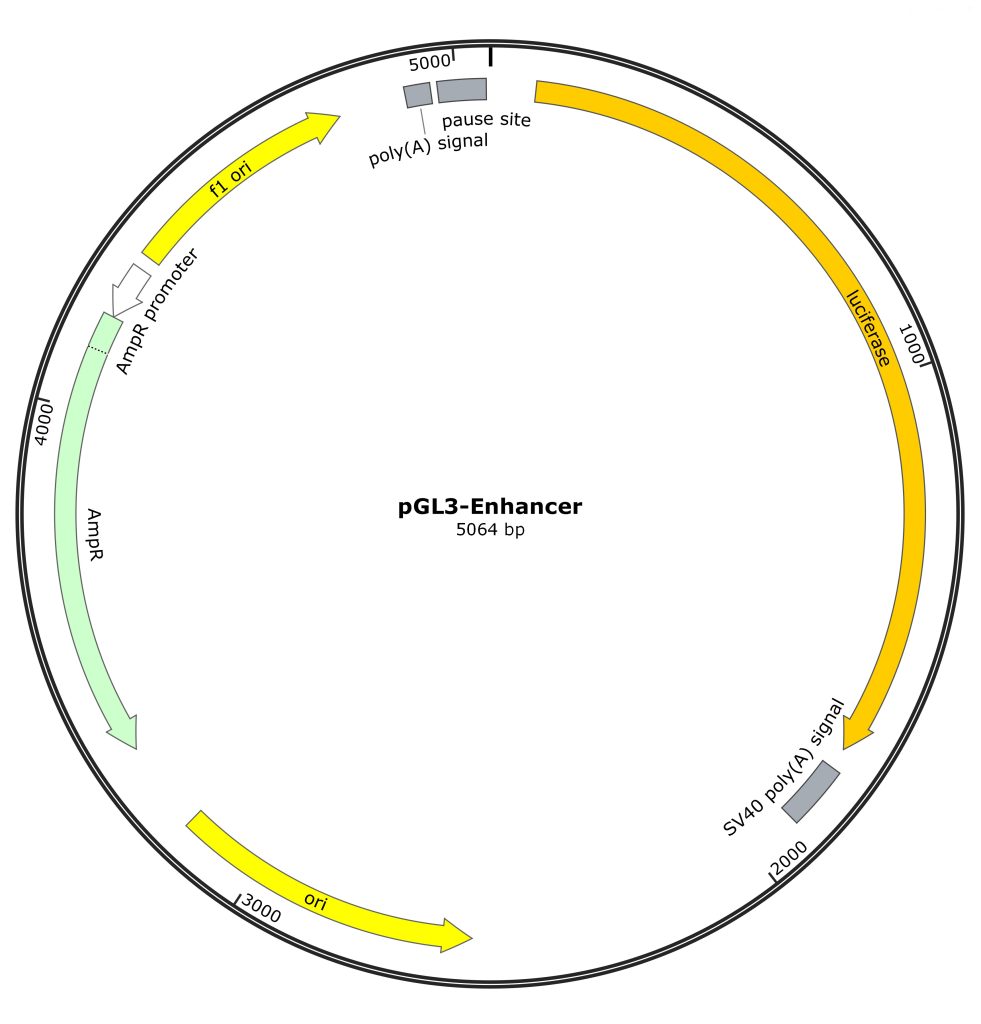
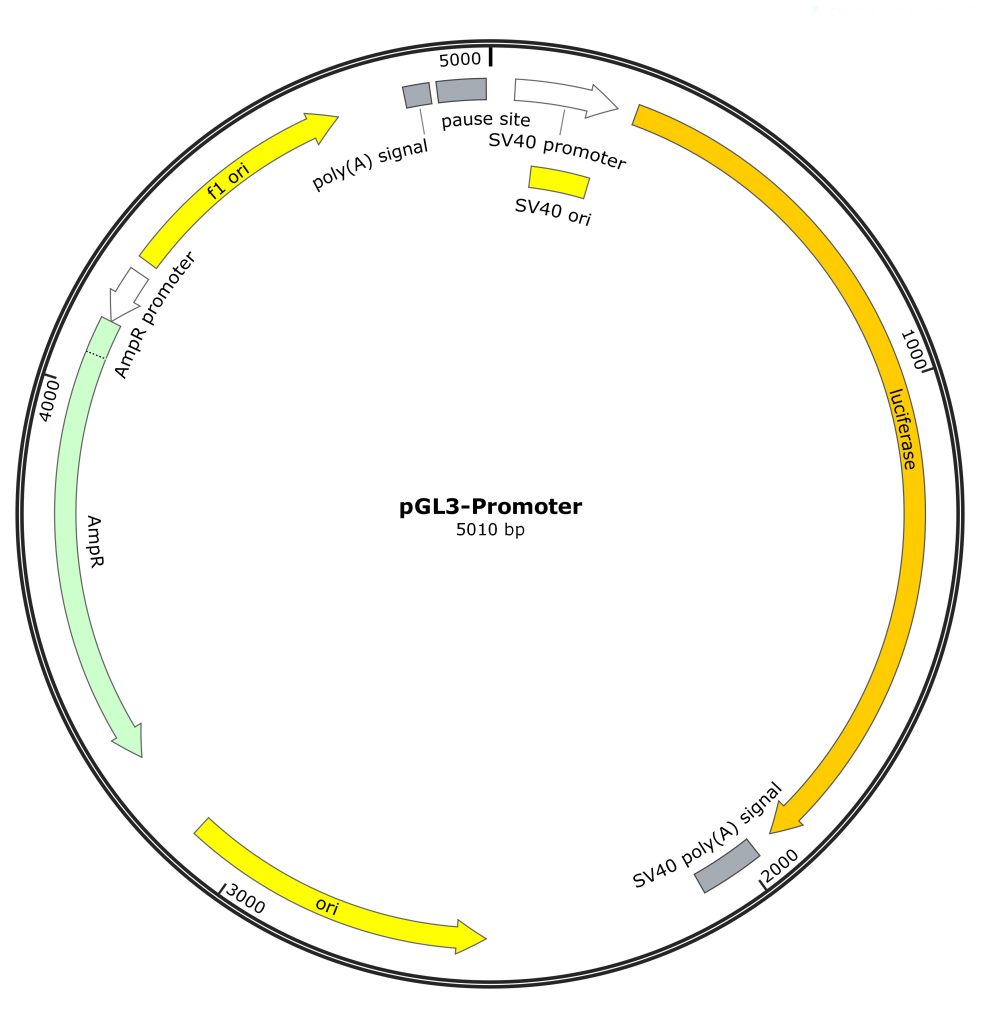
PGL3系列载体包括Promoter、Enhancer、Basic以及Control,都含有萤火虫荧光素酶报告基因,其中Control为对照载体;Basic仅含有一个Luc基因,无启动子与增强子;Promoter含有SV40启动子,可用于检测增强子;Enhancer则与之相反,即含有增强子,可用于检测启动子。
图3 pGL3系列载体图谱。
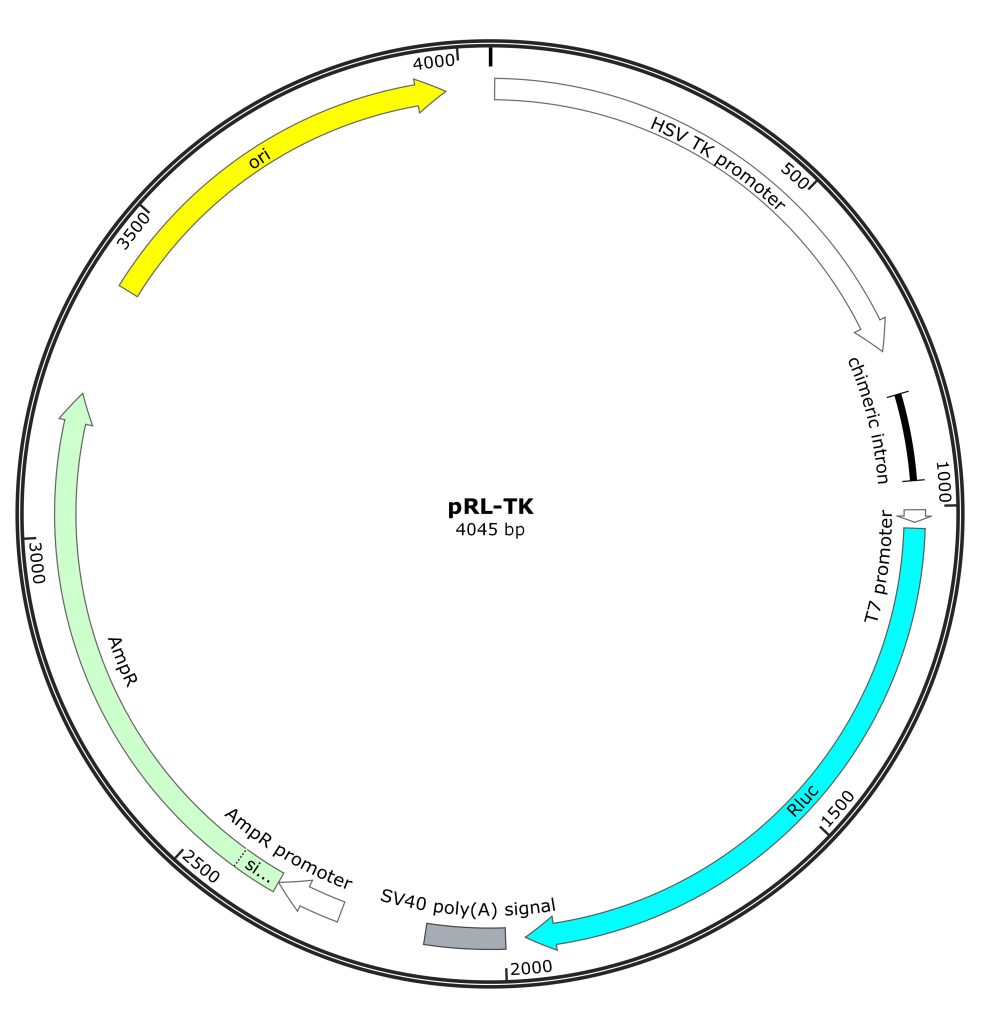
(3)pRL系列载体
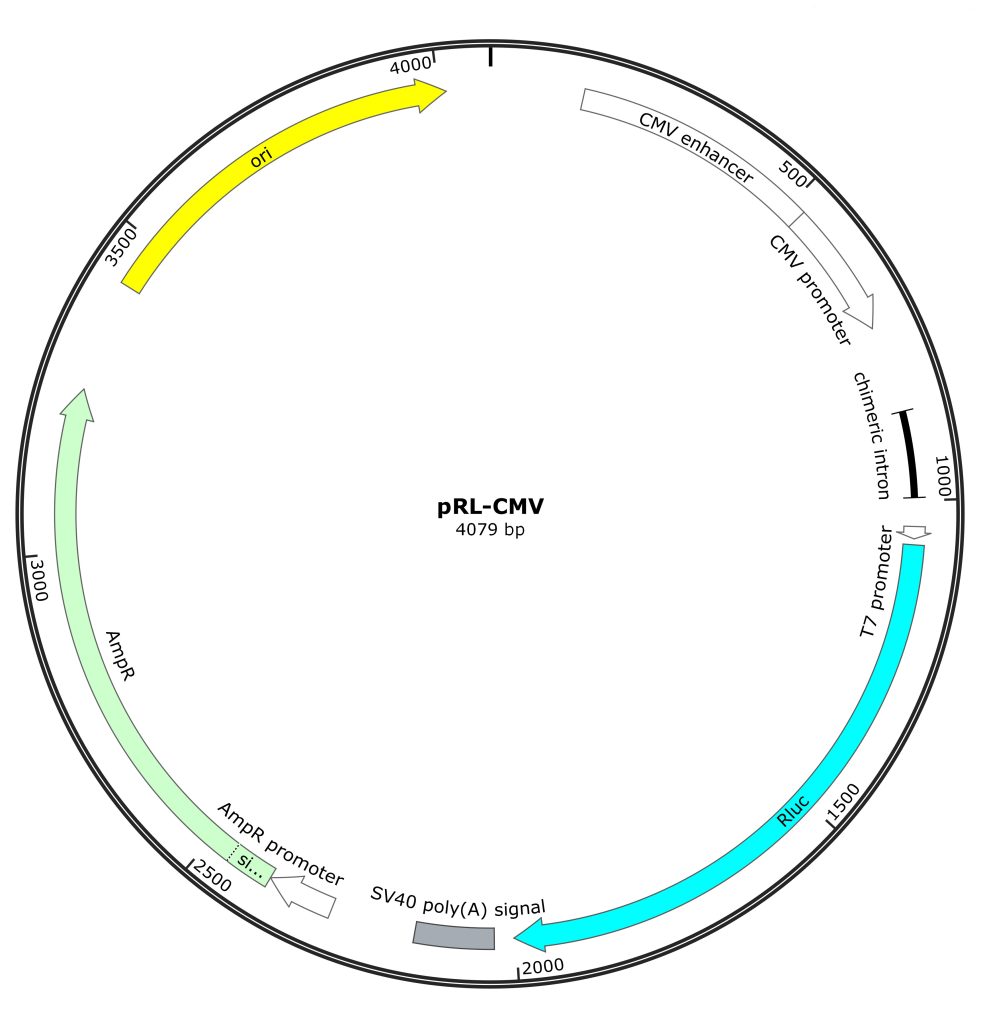
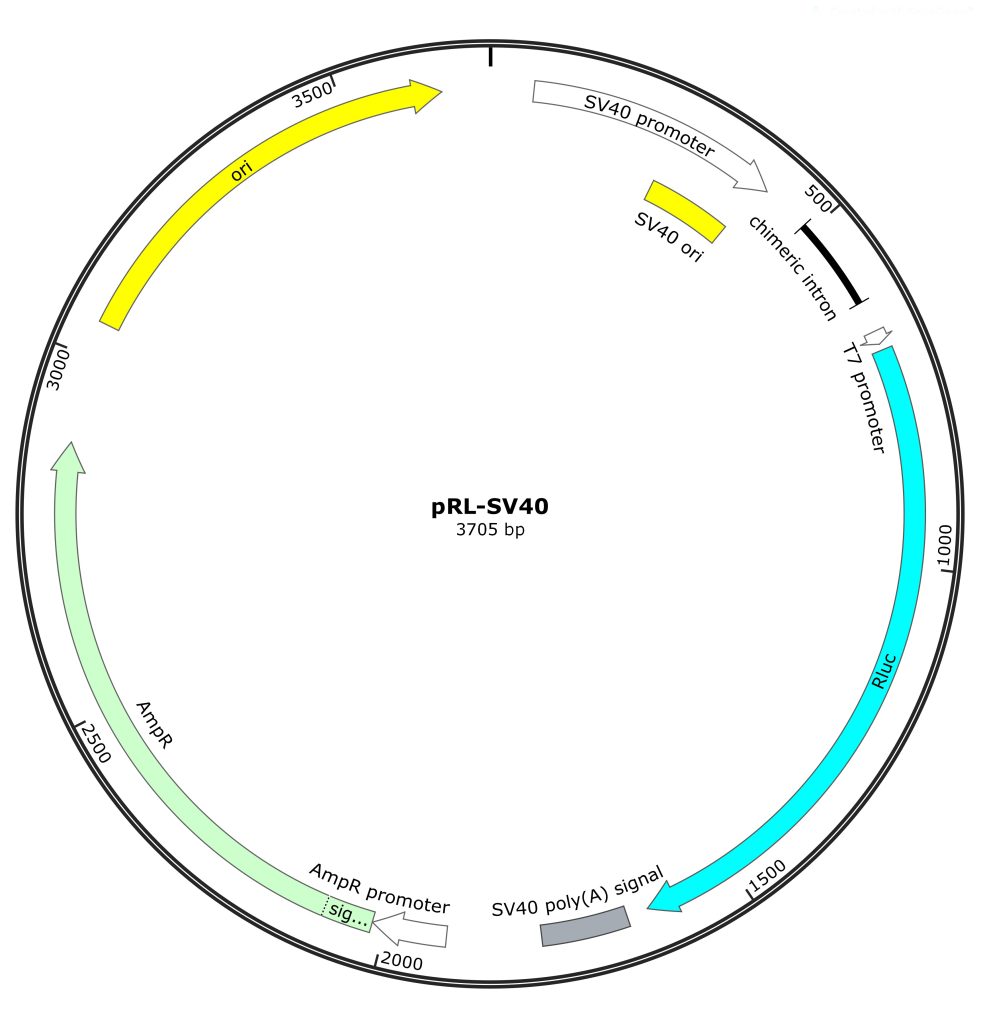
pRL系列载体为海肾荧光素酶报告载体,由Promega公司开发,其载体间最主要的区别是携带的启动子不同,包括CMV、SV40、TK等,即pRL-CMV、pRL-SV40、载体,将F-Luc载体与其共转染293细胞时,pRL系列载体起发挥内参作用。
注:通常,内参基因表达的活性应当显著高于背景组,同时,尽量小,确保不干扰主报告基因的表达。因此,建议选择内参载体时,首先考虑活性较弱的TK启动子驱动的载体。当通过文献资料或者预实验,发现该载体在目标细胞模型中表达效率过低,或者会受到实验研究因素干扰时,再考虑其他表达活性更强的载体,或自己构建所需的特殊启动子载体。
图4 pRL系列载体图谱。
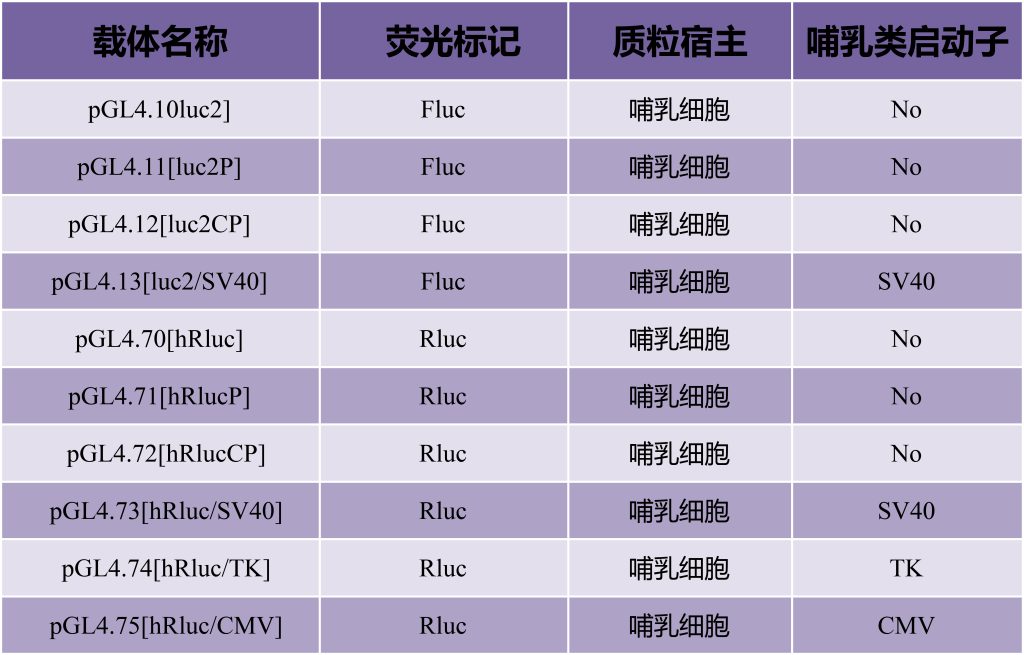
(4)pGL4系列载体
pGL4载体系列代表了新一代萤光素酶报告基因载体,有多种基因和启动子配置供用户选择,其骨架模板就是pGL3载体。具体而言,pGL4系列载体是在pGL3载体骨架的基础上,重新设计了载体中从报告基因起始点到细菌的复制序列的起点,以减少共有转录因子结合位点的数量,从而降低异常表达的风险。不仅如此,pGL4载体的其它修饰还包括重新设计多克隆位点、去除f1复制起始点、去除一个内含子序列及减少启动子数量等等。此外,pGL4系列载体还包含携带优化的海肾萤光素酶编码基因Rluc的载体。
表 pGL4系列载体部分成员
考虑到实验偏差、细胞转染难易程度、细胞承受力等问题,研究人员又进一步对双荧光报告系统的载体进行优化,将两种荧光素酶基因引入同一个载体,以实现偏差小、转染易、细胞死亡率降低的目的。
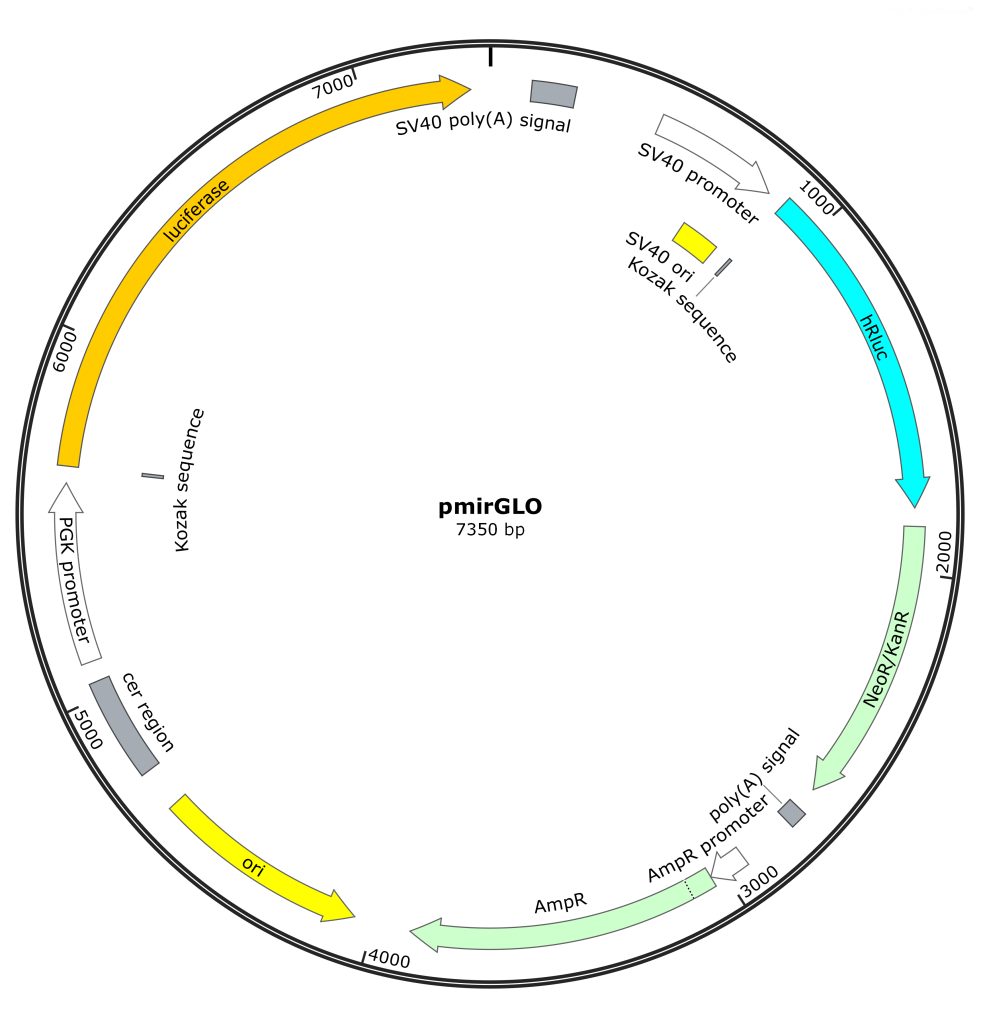
目前,最常用的是Promega公司研发的pmirGLO载体,以PGK中等强度启动子启动的萤火虫萤光素酶基因(luc2)为主报告基因,以海肾萤光素酶基因(hRluc-neo)作为内参报告基因,载体上带有抗性基因,可作为稳定转染筛选标记。
依据检索到的文献显示,现在的这类载体是应用最多的载体,也是我司进行双荧光素酶报告实验推荐载体。
图5 pmirGLO载体图谱。
(2)psiCHECK系列载体
psiCHECK系列载体最初是为验证siRNA靶标而设计的,但介于其是以R-Luc为主报告基因,而F-luc作为内参报告基因,与大部分实验者的使用习惯相悖,且酶切位点少,SV40启动子活性高,不适用于miRNA相关研究。因此,此处不做过多说明,感兴趣的小伙伴可自行学习。
答:研究启动子相关的目的片段构建在萤火虫荧光素酶基因的上游,研究microRNA与mRNA互作的相关片段构建在下游。
(2)可以用萤火虫荧光素酶基因做内参吗?
答:一般以海肾荧光素酶基因做内参,以萤火虫荧光素酶基因做内参也可以(如:psiCHECK系列载体),对应的目的片段构建在海肾荧光素酶基因的上下游就行。
(3)miRNA是构建在载体上表达,还是化学合成?
答:都可以
目前,研究启动子功能应用较为广泛的方法为报告基因研究,将待测启动子构建至荧光素酶基因的上游,检测启动子的活性。
图6 我司验证启动子活性时的载体构建原理图。图片来源:伯远生物科研绘图团队。
文献举例
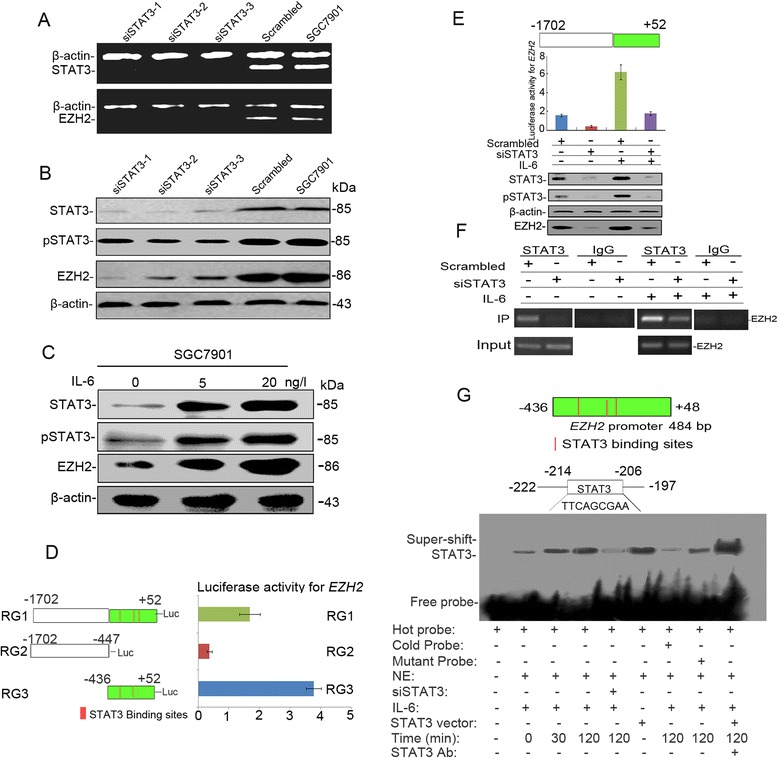
2016年12月9日,Pan等人在Molecular Cancer杂志上发表了题为“STAT3 signaling drives EZH2 transcriptional activation and mediates poor prognosis in gastric cancer.” 的研究论文,在该文中作者首先通过免疫组织化学染色法(IHC)检测了胃癌(GC)患者样本中STAT3、p-STAT3和EZH2的表达,发现它们均过度激活,且与晚期TNM分期和预后不良相关;随后通过双荧光素酶报告基因分析结合EMSA、ChIP分析,确定了EZH2表达是否受STAT3直接调控,STAT3介导EZH2上调。
采用siSTAT3转染SGC7901细胞后,发现STAT3 siRNAs降低了STAT3和EZH2的表达水平,而IL-6刺激可诱导高水平的STAT3和EZH2(图7A-C)。之后,用不同荧光素酶报告载体(包含全长启动子(区域1)、仅包含STAT3结合位点的区域(区域3)、不包含结合位点的区域(区域2))转染SGC7901细胞,结果在包含STAT3结合位点的区域1和区域3中检测到EZH2较高活性(图7D);同时,结果还发现,具有全长EZH2启动子(-1702/+52)的双荧光载体在有或没有IL-6刺激的情况下可通过siSTAT3处理而使其失活(图7E)。为保证实验的可靠性,作者同是采用ChIP与EMSA实验对其进行了验证(图7F、G)。
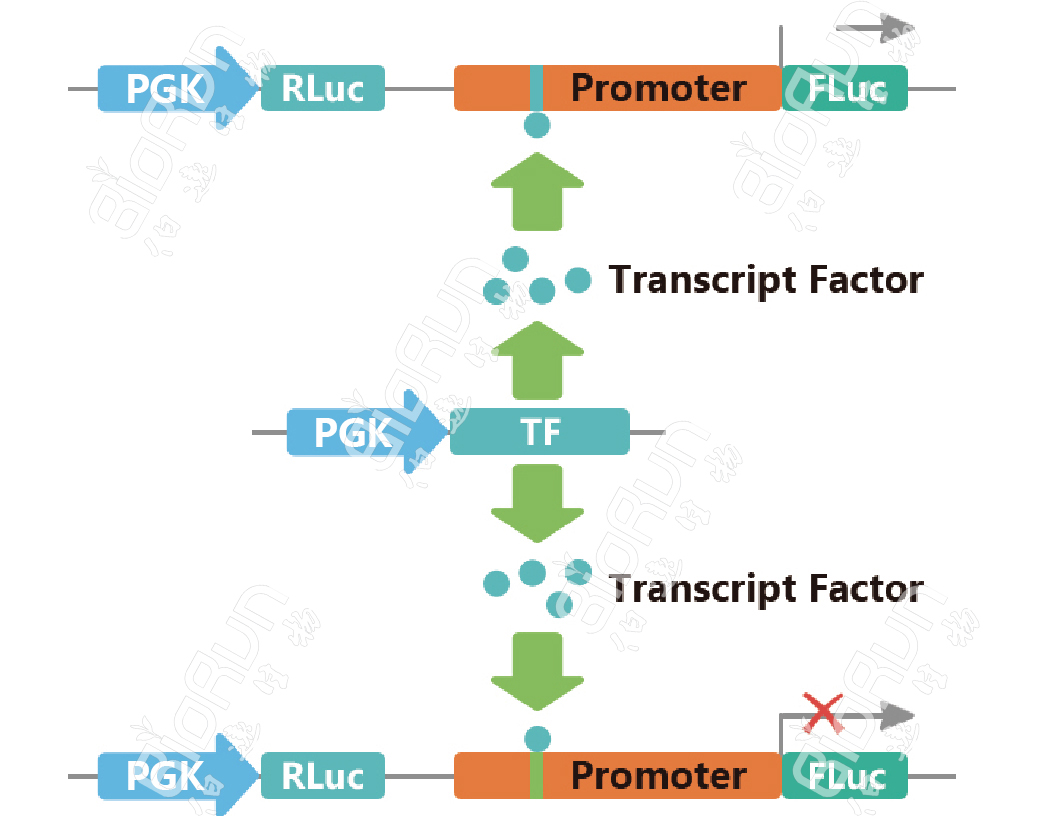
将启动子区域插入双荧光素酶报告基因载体,同时在实验细胞中共转过表达对应的转录因子,可分析转录因子过表达是否提高或降低荧光素酶活性。
图8 我司验证特定转录因子与启动子互作时的载体构建原理图。图片来源:伯远生物科研绘图团队。
文献举例
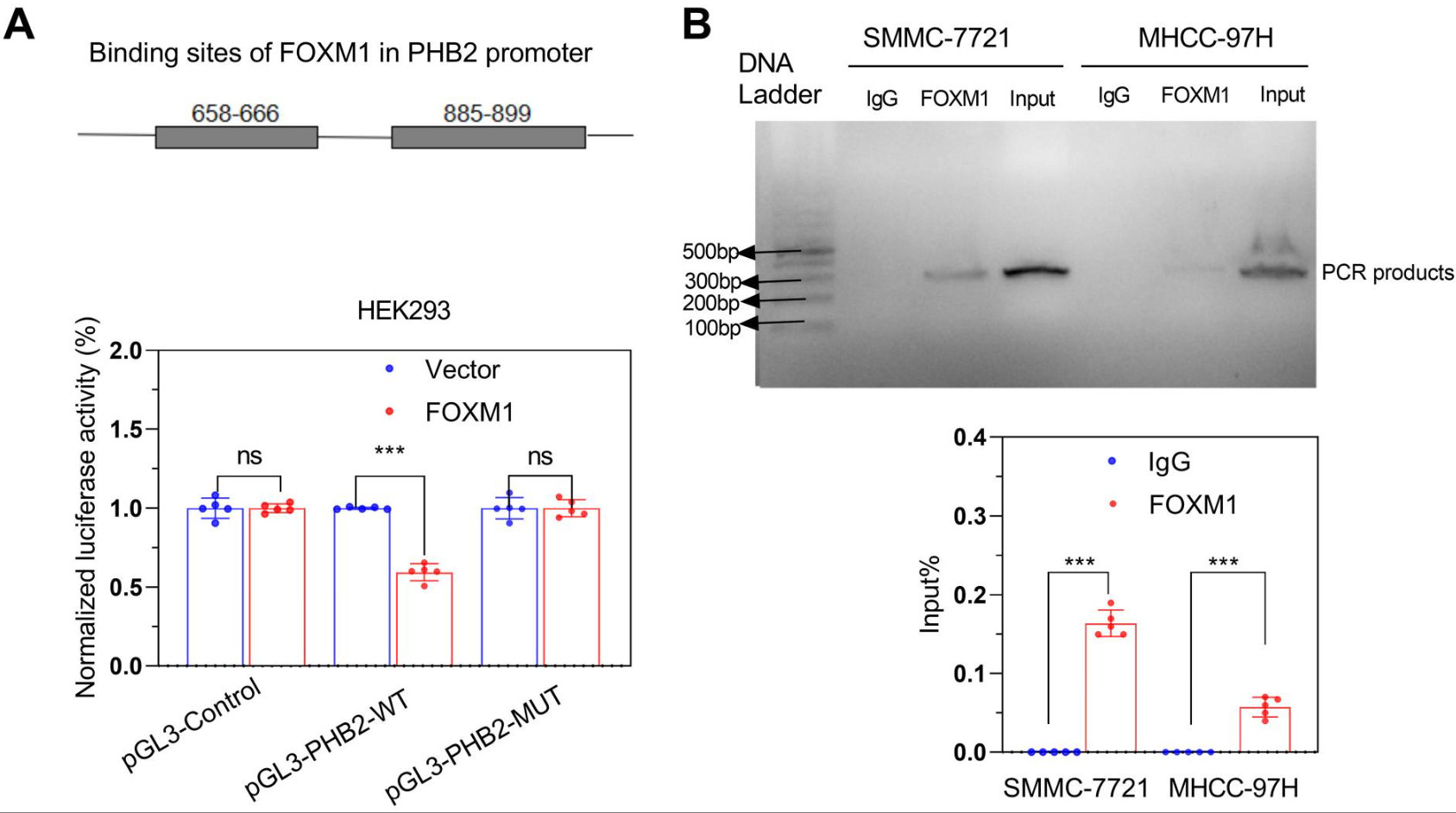
2021年3月4日,Xiang等人在Theranostics杂志上发表了题为“Cellular senescence in hepatocellular carcinoma induced by a long non-coding RNA-encoded peptide PINT87aa by blocking FOXM1-mediated PHB2.”的研究论文,在该文中作者首先通过体内外实验检测了PINT87aa和截短PINT87aa功能,同时结合细胞功能实验进行验证;随后,结构分析预测、免疫共沉淀、免疫荧光共定位证实了PINT87aa与转录因子FOXM1之间可能的结合,并在FOXM1过表达后,进行了体内外回补实验。最后,作者还进行了双荧光素酶报告基因测定与ChIP实验,以验证FOXM1与PHB2启动子的结合(图9)。
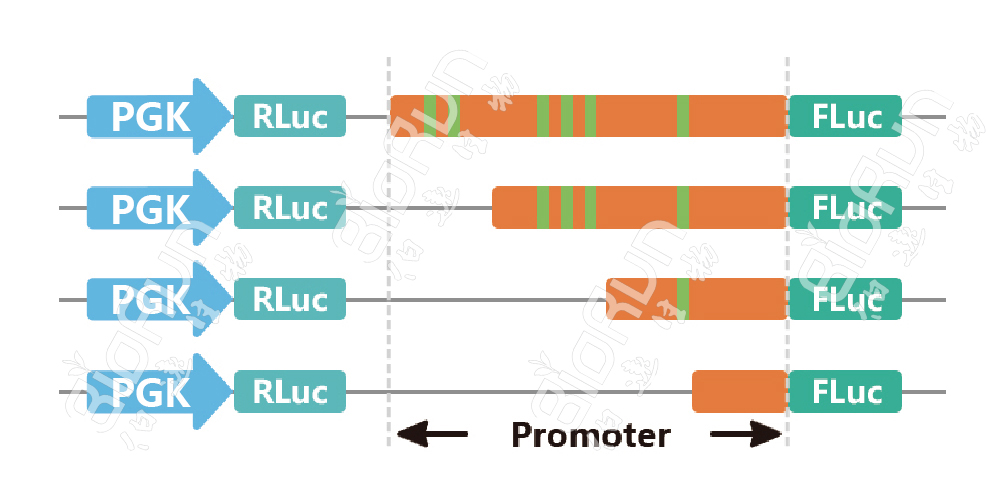
将启动子区域序列(通常2k左右)进行分段截短,或对特定位点进行突变,再分别构建入双荧光素酶报告载体,检测其启动子活性,进而确定启动子中的核心元件。
图10 我司分析启动子结构时的载体构建原理图。图片来源:伯远生物科研绘图团队。
文献举例
2013年10月16日,Qu等人在PLOS ONE杂志上发表了题为“Different involvement of promoter methylation in the expression of organic cation/carnitine transporter 2 (OCTN2) in cancer cell lines.”的研究论文,在该文中作者研究了CpG岛甲基化作为导致癌细胞系中OCTN2下调的可能机制,通过使用甲基化特异性PCR、亚硫酸氢盐基因组测序和体外甲基化测定,发现启动子DNA甲基化是抑制癌细胞系中OCTN2表达的重要机制的证据,应用去甲基化试剂可调节OCTN2启动子的甲基化状态,促进OCTN2表达,并致使癌细胞对奥沙利铂(一种抗癌药)更敏感。
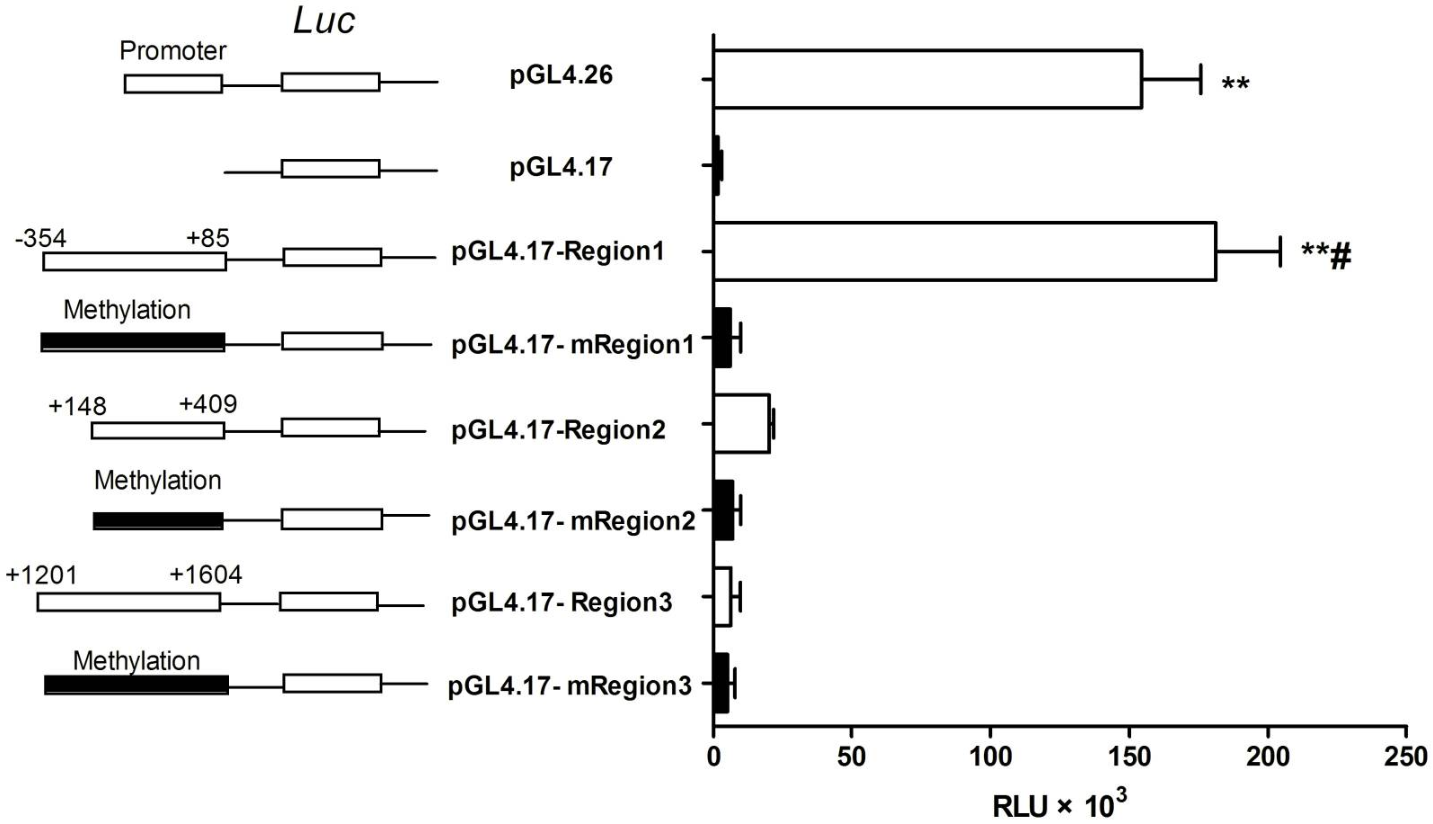
为了确定哪些甲基化CpG岛在OCTN2的下调中起重要作用,作者分别构建了带有未甲基化或甲基化区域的荧光素酶报告质粒,将构建的质粒转染到COS-7细胞中并用于评估转录活性,与对照相比,pGL4.17-Region1的荧光素酶活性显著增加,表明-354-+85 bp的区域在启动子活性中发挥重要作用(图11)。
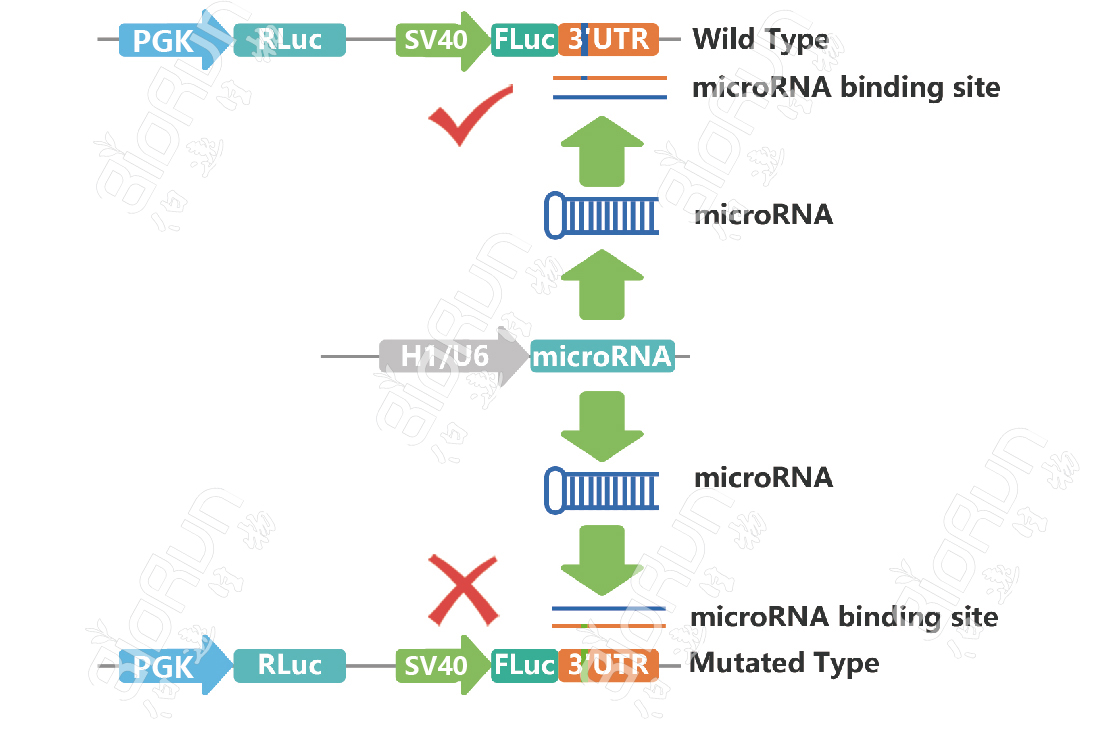
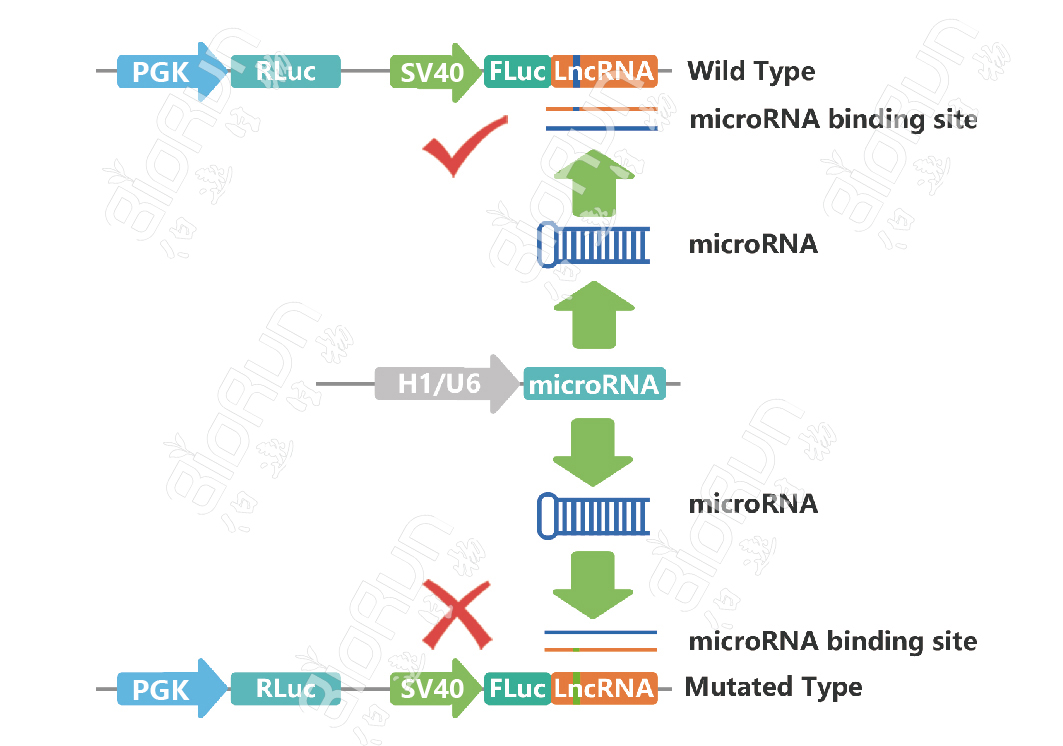
将待测mRNA的3’UTR序列插入报告基因载体,再共转入对应的miRNA,如果荧光素酶活性下降,则提示为其靶序列,同理,这也可用于验证miRNA同lncRNA之间的靶向互作。
图12 我司验证miRNA与mRNA靶向互作时的载体构建原理图。图片来源:伯远生物科研绘图团队。
图13 我司验证miRNA与lncRNA靶向互作时的载体构建原理图。图片来源:伯远生物科研绘图团队。
2019年10月15日,Chen等人在American Journal of Translational Research杂志上发表了题为“LINC01234 promotes multiple myeloma progression by regulating miR-124-3p/GRB2 axis.”的研究论文,在该文中作者通过临床数据评估确认lncRNA LINC01234可能在多发性骨髓瘤(MM)中发挥重要作用。接下来,作者通过细胞功能性实验验证以及体内裸鼠皮下成瘤实验,结合RNA-FISH、RIP、RNA pull down、双荧光素酶报告基因测定确定基于抗增殖、抗细胞周期进展和促凋亡的功能,LINC01234/miR-124-3p/GRB2信号轴可能有望成为MM治疗的有希望的候选者。
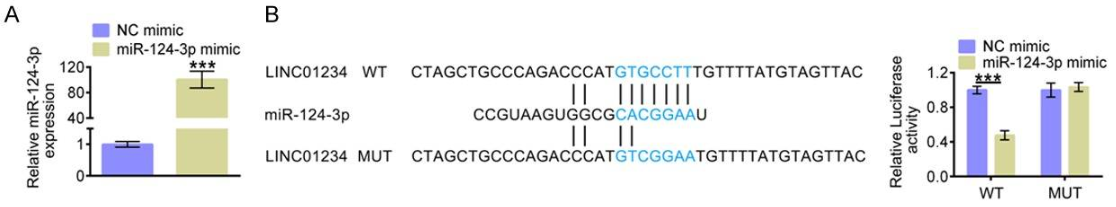
为了探索LINC01234潜在靶miRNA,作者通过miRDB在线数据库预测分析发现,LINC01234可能与miR-124-3p相互作用,转染miR-124-3p mimic后miR-124-3p水平显著上调(图14A);接下来,又通过StarBase在线数据库确定了miR-124-3p与LINC01234之间存在的潜在结合位点(图14B),并通过双荧光素酶报告基因检测实验进行了验证,结果证明,在U266细胞中,miR-124-3p上调抑制LINC01234报告质粒的荧光素酶活性,而在miR-124-3p结合位点突变后没有显著影响(图14B)。
图14 双荧光素酶报告基因实验验证LINC01234与miR-124-3p结合位点(Chen et al., 2019)。
随后,作者通过TargetScan、miRDB和starBase在线数据库预测了miR-124-3p的靶基因,发现GRB2可能是miR-124-3p的潜在靶标(图15A);在确定miR-124-3p与GRB2之间的潜在结合位点后(图15B),通过双荧光素酶报告基因检测实验进行了验证,结果证明,在RPMI-8226或U266细胞中,miR-124-3p上调降低了GRB2 3’UTR报告质粒的荧光素酶活性,而miR-124-3p结合位点突变后荧光素酶活性没有显著影响(图14B)。
图15 双荧光素酶报告基因实验验证miR-124-3p与其靶基因GRB2的结合位点(Chen et al., 2019)。
Pan YM, Wang CG, Zhu M, et al. STAT3 signaling drives EZH2 transcriptional activation and mediates poor prognosis in gastric cancer. Mol Cancer. 2016;15(1):79.
Qu Q, Qu J, Zhan M, et al. Different involvement of promoter methylation in the expression of organic cation/carnitine transporter 2 (OCTN2) in cancer cell lines. PLoS One. 2013;8(10):e76474.
Xiang X, Fu Y, Zhao K, et al. Cellular senescence in hepatocellular carcinoma induced by a long non-coding RNA-encoded peptide PINT87aa by blocking FOXM1-mediated PHB2. Theranostics. 2021;11(10):4929-4944.
赵斯斯. 家蚕P450基因CYP9A19和CYP9A22的启动子分析. 苏州: 苏州大学, 2012.
张菊梅, 吴清平, 周小燕, 等. 荧光素酶研究进展. 微生物学通报, 2001, 28(5): 98-101.
| 官网链接:medicine.biorun.com |