题目
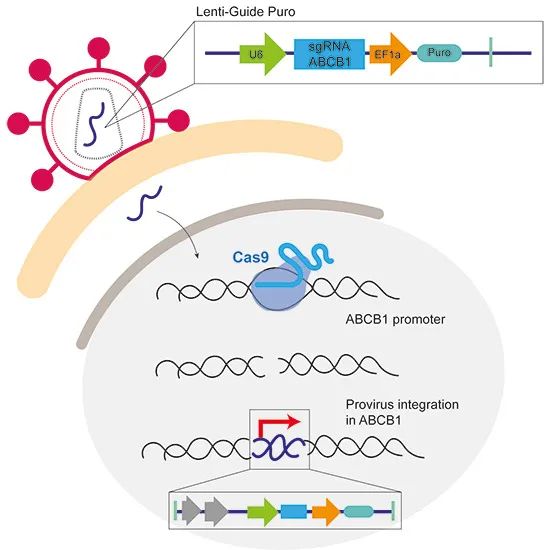
The discovery of the Clustered Regularly Interspaced Short Palindromic Repeats (CRISPR) and its development as a genome editing tool has revolutionized the field of molecular biology. In the DNA damage field, CRISPR has brought an alternative to induce endogenous double-strand breaks (DSBs) at desired genomic locations and study the DNA damage response and its consequences. Many systems for sgRNA delivery have been reported in order to efficiently generate this DSB, including lentiviral vectors. However, some of the consequences of these systems are not yet well understood. Here, we report that lentiviral-based sgRNA vectors can integrate into the endogenous genomic target location, leading to undesired activation of the target gene. By generating a DSB in the regulatory region of the ABCB1 gene using a lentiviral sgRNA vector, we can induce the formation of Taxol-resistant colonies. We show that these colonies upregulate ABCB1 via integration of the EEF1A1 and the U6 promoters from the sgRNA vector. We believe that this is an unreported CRISPR/Cas9 on-target effect that researchers need to be aware of when using lentiviral vectors for genome editing.

